超级增强子研究这么热,你知道怎么鉴定吗?
超级增强子富集在基因组的变异区,而这些变异区与多种疾病谱系密切相关,所以它们最终可能在疾病诊断、预后判断和治疗方面发挥重要作用。超级增强子可作为一种有用的生物标记和治疗靶标,鉴定并干预疾病相关超级增强子,将有望促进精准医学的发展,带来疾病诊断和治疗的技术变革。
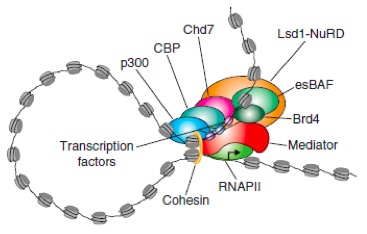
图1.超级增强子及复合物结构示例图[1]
目前对增强子鉴定,主要采用染色质免疫共沉淀技术(ChIP-seq) 针对活性增强子相关联的因子或组蛋白修饰进行检测,如转录因子、转录辅激活因子(如Mediator、p300)、组蛋白修饰H3K27ac 和H3K4me1等。活性增强子通常同时含有H3K27ac 和H3K4me1 修饰,而静态增强子(poised enhancer)一般同时具有H3K4me1 和H3K27me3 组蛋白标记[4-6]。在此基础上,超级增强子依据增强子转录活性标记分子结合水平强度的差异进行鉴定。在分析方法上,首先对所得增强子进行缝合。主要依据在基因组范围内,这些单个增强子实体间如在12.5 kb 范围内,则合并为单个实体,即缝合增强子(Stitched enhancer)。最后,确定超级增强子和普通增强子之间的阈值。缝合增强子和其余的单个增强子按照ChIP-seq 所测信号水平的强度排序,绘制获得一张曲线图,该曲线上斜率为1 的切线的切点所得的信号值为区分超强增强子和普通增强子之间的阈值,高于该值为超级增强子,其余的则称为普通增强子(Typical enhancer)[1,2](见图2,图3)
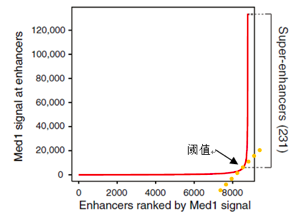
图2. 确定超级增强子和普通增强子之间的阈值[1]
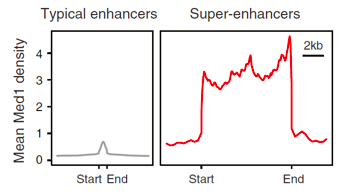
图3.普通增强子与超级增强子示例图[3]
鉴定出超级增强子之后,可以根据基因位置预测超级增强子所调控的编码蛋白基因和非编码RNA的表达,通过结合转录组测序技术,可以对超级增强子和肿瘤异常高表达的mRNA、lncRNA、circRNA、miRNA进行关联分析,从而推断关键的超级增强子,进一步锁定肿瘤相关的基因,有利于指导下一步的抗肿瘤功能研究。
癌症细胞可以通过突变、关键正常基因超级增强子的染色体易位、局部扩增、过度表达致癌转录因子等遗传机制来构建驱动致癌基因的超级增强子[3,7,8]。目前研究表明常见的复杂疾病和超级增强子之间也是相关联的。Richard A. Young 实验室对GWAS 鉴定的5 303 个疾病特征相关SNP 分布分析表明,大部分SNP 仅生在非编码区域(93%),其中64%的位点富集于增强子区域,并且富集于超级增强子的变异要显著多于普通增强子[3](见图4)。因此锁定目标超级增强子区域后,可以结合大样本的靶向捕获测序,对目标增强子区域进行DNA测序,有助于快速发现肿瘤相关的突变位点,获得新的诊断标志物。

图4. 超级增强子区域存在大量疾病相关的突变位点[3]
表观生物技术团队具备丰富的ChIP实验经验,利用经验证的ChIP级抗体,提供高质量ChIP-seq技术和生物信息分析,倾力打造国内领先超级增强子鉴定服务整体解决方案。
项目流程 :
样本要求:
仅限人及大鼠或小鼠细胞和组织
细胞量达到107个
测序模式:
SE50
测序数据量:
20M
生物信息分析内容
一、标准信息分析
1. 去接头污染,去低质量reads和测序质量评估
2. ChIP测序序列与参考基因组序列的比对
3. ChIP测序唯一reads在全基因组的分布
4. Peak鉴定及基因原件分析
5. Peak相关基因筛选与GO功能聚类分析、Pathway分析
二、高级信息分析
1、普通增强子与超级增强子鉴定分析;
2、普通增强子与超级增强子差异(长度及数目)分析;
3、超级增强子鉴定及数量分析图绘制;
4、普通增强子与超级增强子信号富集图绘制;
5、超级增强子的注释及调控靶基因预测;
6、超级增强子调控靶基因KEGG、GO分析;
7、超级增强子结合转录因子预测
参考文献
[1] Whyte WA, Orlando DA, Hnisz D, Abraham BJ, Lin CY, Kagey MH, Rahl PB, Lee TI, Young RA. Master transcription factors and mediator establish super-enhancers at key cell identity genes. Cell, 2013, 153(2): 307–319.
[2] Lovén J, Hoke HA, Lin CY, Lau A, Orlando DA, Vakoc CR, Bradner JE, Lee TI, Young RA. Selective inhibition of tumor oncogenes by disruption of super-enhancers. Cell, 2013, 153(2): 320–334.
[3] Hnisz D, Abraham BJ, Lee TI, Lau A, Saint-André V, Sigova AA, Hoke HA, Young RA. Super-enhancers in the control of cell identity and disease. Cell, 2013, 155(4): 934–947.
[4] Creyghton MP, Cheng AW, Welstead GG, Kooistra T,Carey BW, Steine EJ, Hanna J, Lodato MA, Frampton GM,Sharp PA, Boyer LA, Young RA, Jaenisch R. Histone H3K27ac separates active from poised enhancers and predicts developmental state. Proc Natl Acad Sci USA, 2010,107(50): 21931–21936.
[5] Heintzman ND, Hon GC, Hawkins RD, Kheradpour P,Stark A, Harp LF, Ye Z, Lee LK, Stuart RK, Ching CW, Ching KA, Antosiewicz-Bourget JE, Liu H, Zhang XM,Green RD, Lobanenkov VV, Stewart R, Thomson JA,Crawford GE, Kellis M, Ren B. Histone modifications at human enhancers reflect global cell-type-specific gene expression. Nature, 2009, 459(7243): 108–112.
[6] Rada-Iglesias A, Bajpai R, Swigut T, Brugmann SA, Flynn RA, Wysocka J. A unique chromatin signature uncovers early developmental enhancers in humans. Nature, 2011,470(7333): 279–283.
[7] Mansour MR, Abraham BJ, Anders L, Berezovskaya A,Gutierrez A, Durbin AD, Etchin J, Lawton L, Sallan SE,Silverman LB, Loh ML, Hunger SP, Sanda T, Young RA,Look AT. Oncogene regulation. An oncogenic super-enhancer formed through somatic mutation of a noncoding intergenic element. Science, 2014, 346(6215):1373–1377.
[8] Gröschel S, Sanders MA, Hoogenboezem R, de Wit E,Bouwman BA, Erpelinck C, van der Velden VHJ, Havermans M, Avellino R, van Lom K, Rombouts EJ, van DuinM, Döhner K, Beverloo HB, Bradner JE, Döhner H, Löwenberg B, Valk PJM, Bindels EMJ, de Laat W, Delwel R.A single oncogenic enhancer rearrangement causes concomitant EVI1 and GATA2 deregulation in leukemia. Cell, 2014, 157(2): 369–381.
- - - 推荐阅读 - - -

Cell案例| lncRNA研究无从下手?你试过这些方法吗
lncRNA TERRA有着高度重复的序列,缺乏合适的功能研究工具。我们来看看来自美国霍华德休斯医学研究中心的研究者是如何解决这个问题的。
2017-08-10

【推荐】RNA甲基化修饰(m6A)研究技术及方案设计
RNA甲基化是表观遗传学内容的重要内容之一,其中m6A(N6-methyladenosine,6-甲基腺嘌呤)较为常见的一种修饰方式。m6A是一种动态可逆的修饰方式,在转录后调控中发挥作用,其在调控基因表达、剪接、RNA 编辑、RNA 稳定性、控制mRNA寿命和降解、介导环状RNA翻译[1]等方面扮演重要角色,具有重要的研究意义.
2017-06-29

【推荐】肿瘤相关超级增强子的研究方案及应用前景
增强子是调控细胞基因时空表达关键的顺式作用元件。2013年,Richard A. Young 实验室基于当时增强子的研究,提出了超级增强子(Super-enhancers, SEs)概念.
2017-06-22
