GRO-seq(新生RNA-seq)服务
项目简介
GRO-seq (Global nuclear Run-On sequencing)是一种专门测量新生RNA的方法,可用于系统鉴定eRNA分子。该方法通过对细胞进行迅速冷冻,使细胞核内正在转录的复合体处于冻结状态,然后提取细胞核,加入Run on缓冲液,使其重新恢复RNA的转录。利用抗BrdU抗体提取带BrU标记的新生成RNA ,用于下一步高通量测序分析。
新生RNA的变化直接反映了转录调控的多种机理,通过GRO-seq技术,可以观测到基因组水平正在进行的转录,这是一种对所有参与RNA聚合酶位置、数量和定向的高分辨率虚拟图谱。

实验流程
测序方案
Illumina NextSeq CN500
测序平台
SE 75
测序模式
40 M
数据量
送样要求
1.具备条件的,可以把细胞核分离好液氮速冻并干冰运输。
2. 不具备前期准备条件的,需提供活细胞及配套培养基,常温运输。如需进行细胞前处理,请提供详细说明和相关材料,并收取相应处理费用。
1x10⁷个
细胞数量
人,大小鼠
仅限物种
应有与分析
应用范围
1、取代常规RNA-seq,精准检测实时转录状态的基因表达
2、精确定位基因的转录位置和转录方向
3、发现直接作用的靶标基因
4、发现新的转录本,包括非编码RNA
5、鉴定eRNAs分子
生物信息分析
1. 数据产出统计,对原始测序数据去接头、去低质量 reads、去污染;
2. 测序序列与参考基因组序列的比对,Reads在全转录组的分布;
3. Reads在转录起始位点(TSS)附近的meta分析;
4. 转录暂停基因分析
5. eRNA分析
6. 差异基因分析
7. 差异基因 GO 功能聚类分析及KEGG生物通路富集分析
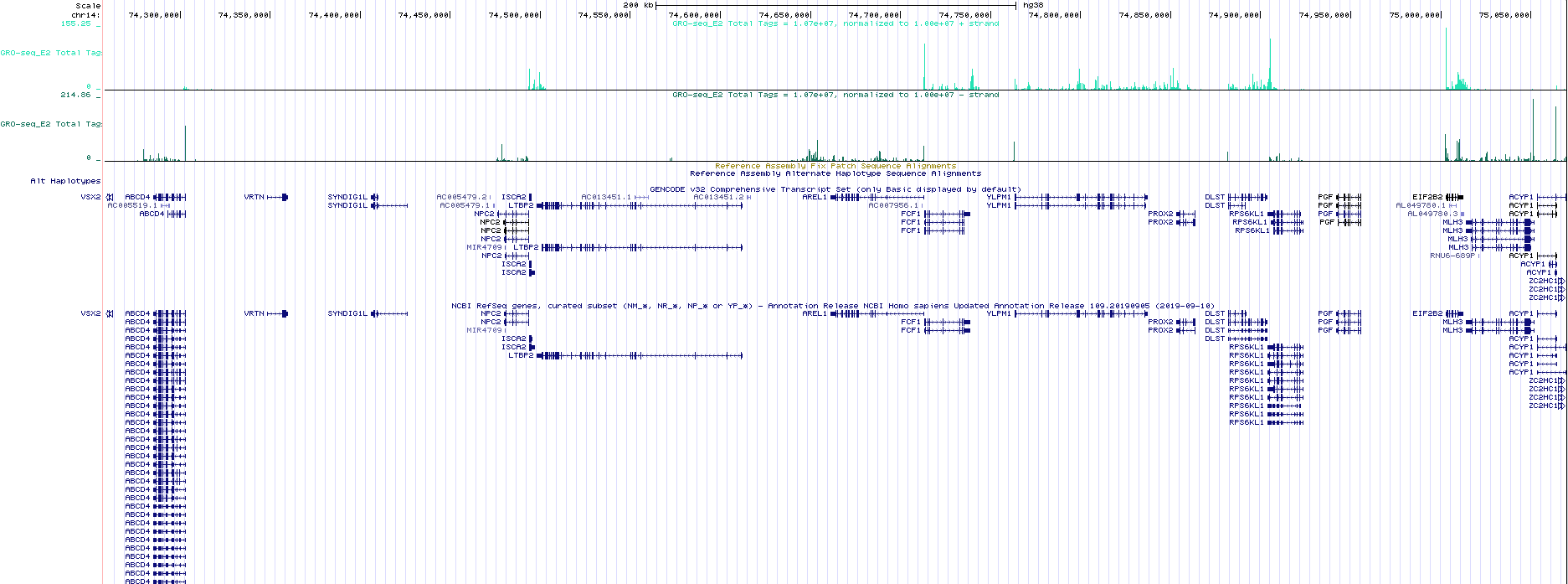
图1. gro-seq数据在UCSC基因组浏览器中可视化bed文件
表观生物实测数据
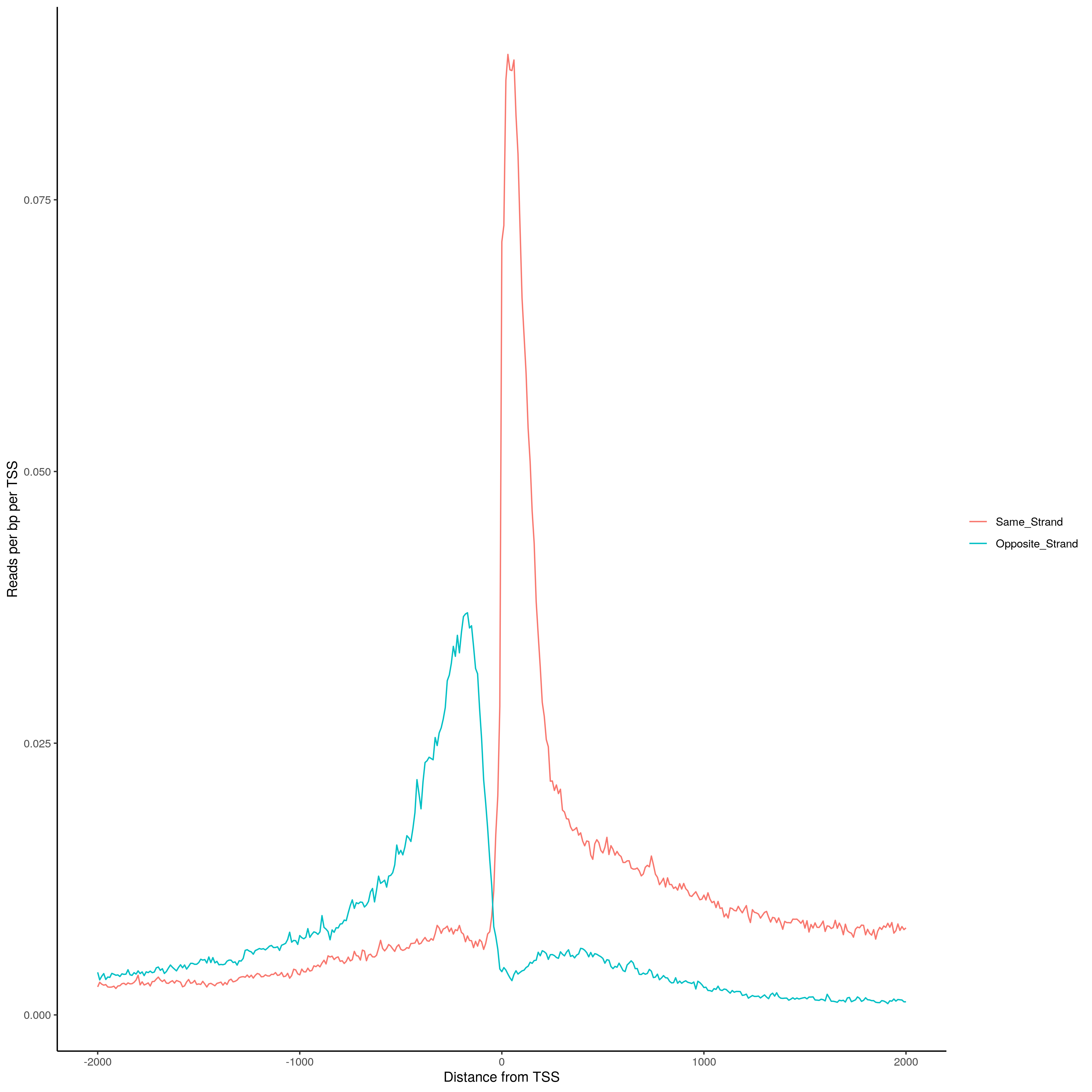
图2. TSS距离直方图
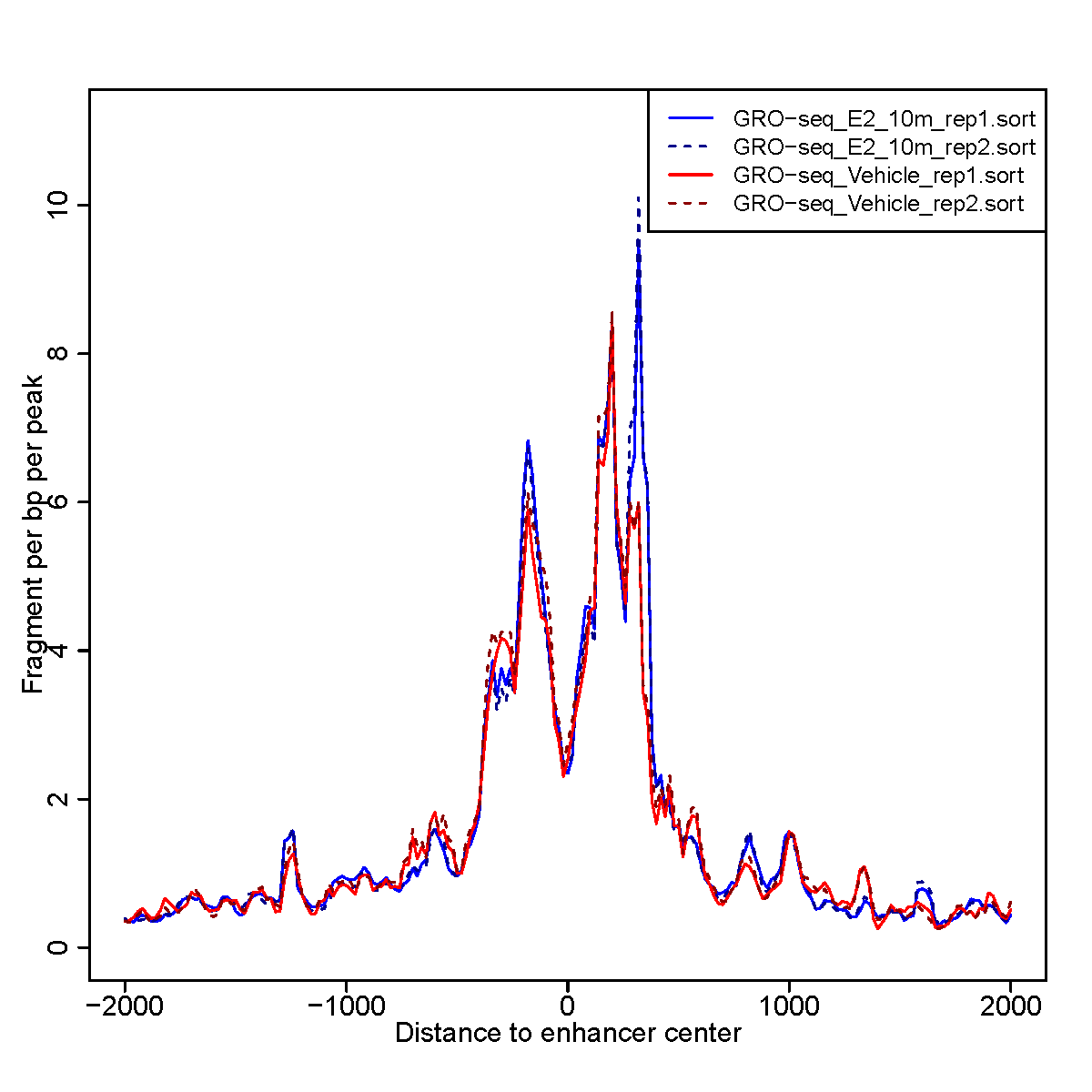
图3. 所有样品均位于增强子中心附近的gro-Seq信号

图4. 两种条件下暂停索引变化箱形图

图5. TSS周围活跃基因的条件依赖性转录变化的热图
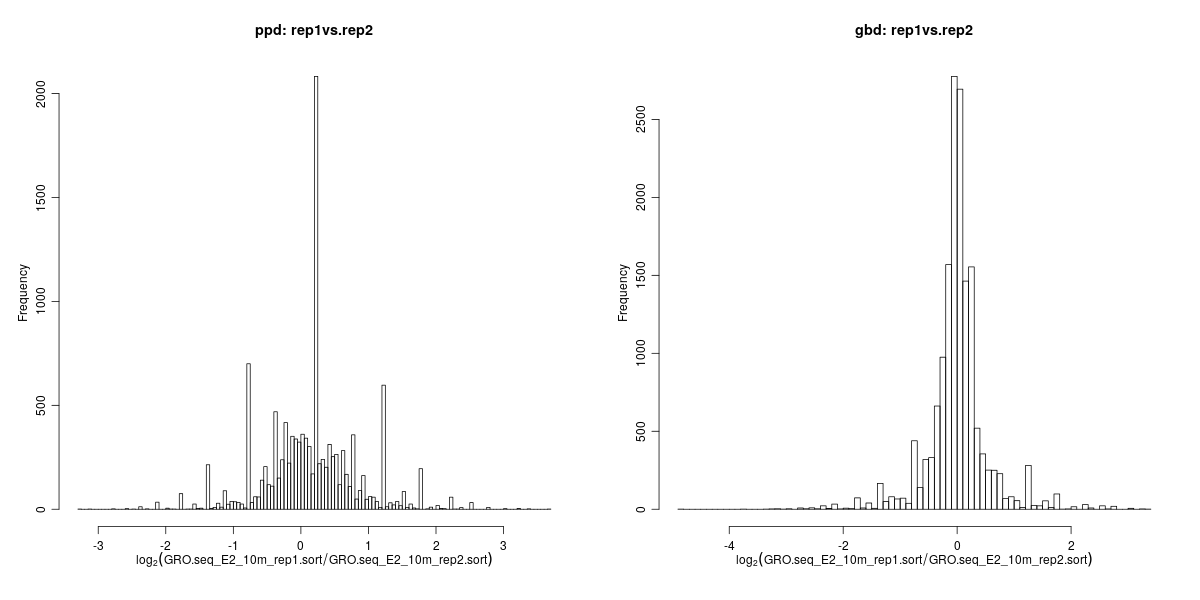
图6. 条件1下重复样本之间转录变化的直方图
参考文献
Core L J, Waterfall J J, Lis J T. Nascent RNA sequencing reveals widespread pausing and divergent initiation at human promoters[J]. Science, 2008, 322(5909): 1845-1848.
Danko C G, Hyland S L, Core L J, et al. Identification of active transcriptional regulatory elements from GRO-seq data[J]. Nature methods, 2015, 12(5): 433-438.
