案例解读 | Genome Biol:空间互作组HiChIP技术助力鉴定乳腺癌风险基因座靶基因
全基因组关联研究(GWAS)已鉴定了超过200个与乳腺癌风险相关的基因座。大多数候选致病突变(Candidate causal variants,或称候选因果变异)位点位于非编码区,可能通过调控基因表达从而介导癌症表型。然而,精确定位大量乳腺癌风险位点上的靶基因并识别其介导的表型是一项艰巨的任务。2023年3月29日,Genome biology(IF=17.906)发表了题为CRISPR screens identify gene targets at breast cancer risk loci[1]的文章,为这个问题的解决提供了一套全新策略。

下面小编将对该文章研究内容进行详细解读~
研究方法
在细胞2D和3D培养物以及小鼠体内进行CRISPR筛选鉴定乳腺癌风险基因,进一步通过HiChIP和CRISPRqtl证实乳腺癌风险基因座与基因的互作;
其中,HiChIP通过结合Hi-C技术和ChIA-PET(空间结构互作)技术,具有所需细胞量少、信噪比高、特异性好等优势;
测序样本:
1.分别用K5+/K19+、K5+/K19- 、B80-T17、mesHMLE、B80-T5、HMLE这六种细胞系进行RNA-seq和ATAC-seq测序;
2.K5+/K19-、B80-T5细胞系用于H3K27ac HiChiP ,研究风险位点与基因之间的互作关系;
研究结果
1.功能性CRISPR筛选基因
研究者利用INQUISIT(整合表达数量性状及其通过计算模型进行GWAS靶基因的预测)、全转录组关联研究(TWAS)和表达数量性状基因座(eQTL)等方法研究鉴定的乳腺癌风险基因[2, 3]。作者设计了960个sgRNAs靶向193个核心基因,还有16个已知的肿瘤抑制基因和原癌基因。
2.在2D和3D细胞培养中筛选鉴定诱导细胞增殖表型的乳腺癌风险基因
细胞增殖受损是癌症的一个标志,研究者通过GWAS确定了205个乳腺癌风险信号上的候选致病突变在基因上的位置,选取六个永生化的乳腺上皮细胞系,抑制(CRISPRko或CRISPRi)以及过表达(CRISPRa)候选乳腺癌风险基因,并鉴定可能的肿瘤抑制基因和癌基因。进一步发现了41个候选乳腺癌风险基因,它们在2D或3D细胞介导了增殖表型。
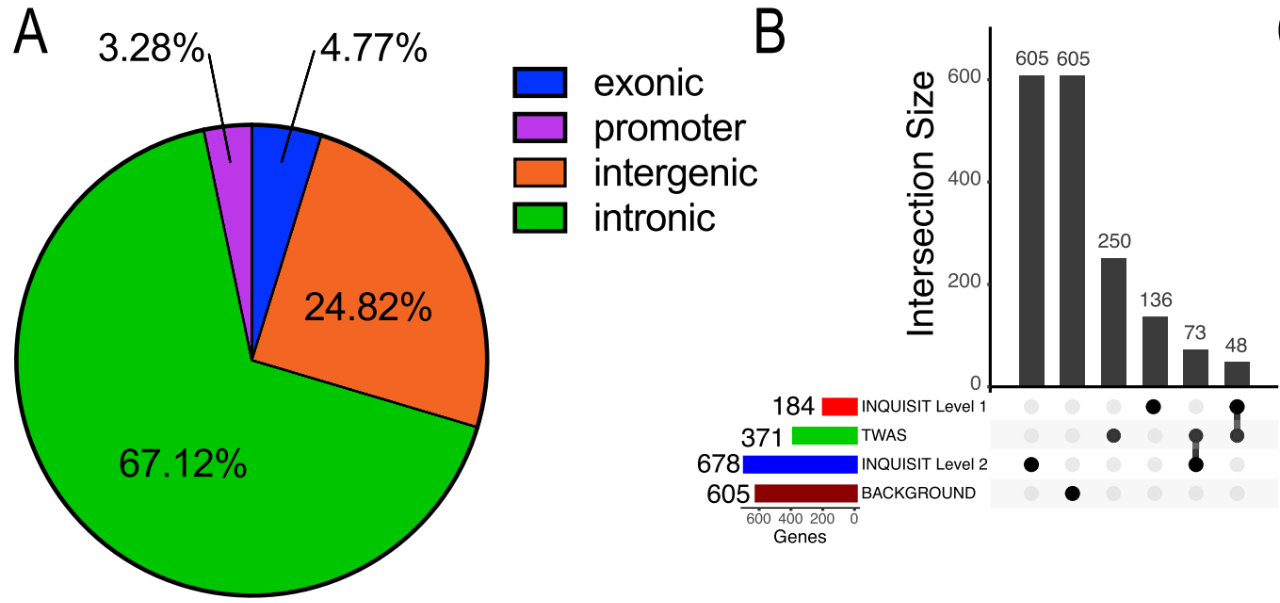
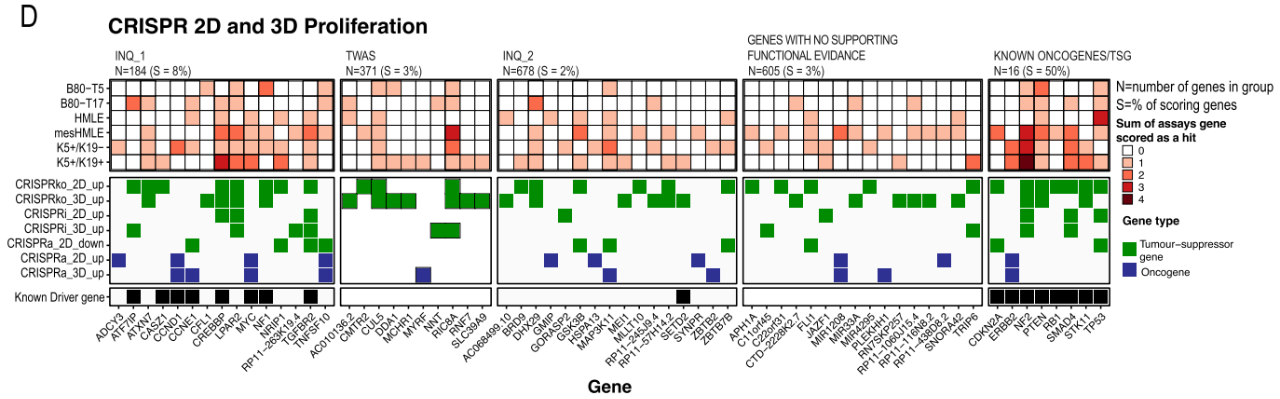
图1 在2D、3D细胞培养物中通过CRISPR激活和抑制筛选鉴定调节细胞增殖的乳腺癌风险基因
3.促进小鼠肿瘤形成的乳腺癌风险基因的鉴定
为了确定在体内肿瘤形成中起作用的候选乳腺癌风险基因,研究者使用了小鼠异种移植模型,5个阳性对照表明sgRNA丰度显著增加,通过INQUISIT预测基因得分揭示了这些基因是乳腺癌风险的有效驱动因素。为了证明DUSP4是否是肿瘤相关抑制因子,作者在B80-T5或B80-T5-MEKDD中敲除DUSP4基因后,测定了3D培养物中的细胞增殖情况。与体内筛选一致,DUSP4基因被敲除后,仅在MEKDD存在时显示细胞增殖能力上升。为了研究DUSP4的作用机制,我们在MEKDD表达后评估了DUSP4的水平,发现MEKDD诱导了DUSP4的表达,暗示了一个负反馈回路。进一步研究者发现MEK抑制剂可以抑制DUSP4蛋白水平的增加以及DUSP4诱导的细胞增殖,表明MEK抑制剂是使乳腺癌DUSP4表达下调的的一种治疗策略。



图2 CRISPR激活和抑制筛选鉴定调节小鼠肿瘤生长的乳腺癌风险基因
通过体内和体外增殖筛选共同确定了44个预测的乳腺癌风险基因,这些基因可以在2D、3D培养物或体内驱动细胞增殖表型。
4.识别调控DNA损伤反应的基因
DNA损伤通常是癌症的一个标志,特别是在乳腺癌中。为了确定调节DNA损伤反应的乳腺癌风险相关基因,研究者使用PARP抑制剂合成致死筛选。在sgRNA感染后,一种有效的PARP1抑制剂olaparib处理细胞,发现只有含有解除调控同源重组DNA损伤修复途径的sgRNA的细胞才对olaparib敏感,通过比较olaparib和DMSO处理的细胞中sgRNA的丰度,研究者鉴定了抑制或激活使细胞对olaparib敏感的基因,通过选取5个永生化乳腺细胞系,筛选了在抑制(CRISPRko或CRISPRi)或过表达(CRISPRa)时调节DNA修复途径的基因。进一步确定了27个调控DNA损伤反应的候选乳腺癌风险基因,其中8个基因是INQUISIT预测得到的。通过GSEA分析揭示了富集基因参与DNA修复途径和细胞周期。随后,研究者在B80-T5细胞验证了这些结果,表明CRISPR介导的基因抑制(已知的DNA损伤相关基因和新鉴定的合成致死基因:SIVA1和CMTR2))在PARP抑制剂的敏感性方面有显著影响。进一步通过各种计算和统计方法预测靶基因的表达情况,发现INQUISIT Level 1方法下的六个细胞系中有五个细胞系的靶基因存在高表达的情况。
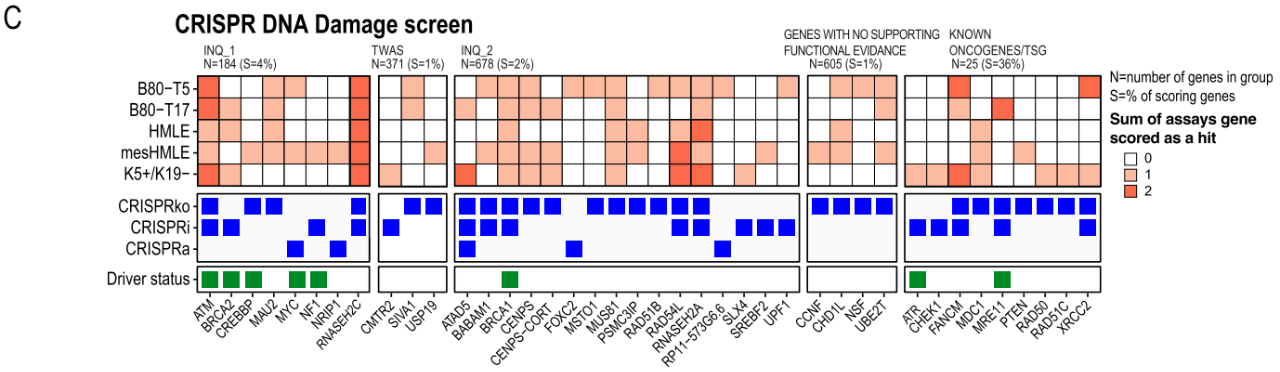

图3 Olaparib合成致死筛选鉴定调控DNA修复途径的乳腺癌风险基因
5.HiChIP测序和CRISPRqtl验证乳腺癌风险的基因座与功能筛选得分的基因之间的远端调控
通过使用CRISPR表型筛选,鉴定了20个基因与其相关的含有乳腺癌风险变异的远端增强子。远端增强子的一种常见机制是通过染色质环(chromatin looping)来调控基因表达。为了在正常乳腺细胞系中证明这一点,通过对B80-T5和K5+/K19+细胞系进行HiChIP测序,发现染色质互作区域包含乳腺癌风险变异。
随后,研究者发现ATF7IP的染色质相互作用特别强,该基因座的风险信号包含18个SNPs,其中7个位于开放染色质和H3K27ac组蛋白标记的候选增强子区域内。向ATF7IP启动子添加含有保护等位基因的ATF7IP假定调控元件(PRE)后,荧光素酶活性增加了9倍(p<0.0001)。这种荧光素酶活性的增加在引入含有风险相关等位基因的PRE后降低了50%(p<0.001)。并且,发现引入rs11055880 (PRE突变体1)的一个变体与整个风险相关等位基因具有相同的效果,而引入rs16909788和rs17221259 (PRE突变体2)对荧光素酶活性没有影响。总的来说,这种影响与该位点的乳腺癌风险相关变异一致,该位点降低了假定的肿瘤抑制基因ATF7IP的表达。由于荧光素酶检测需要外源结构的表达,并且可能不能完全概括原生染色体结构,基于此研究者利用CRISPRi方法进行结果验证。先前研究表明,sgRNAs靶向增强子可有效抑制靶基因的表达。通过使用四个ATF7IP增强子靶向的sgRNAs,发现ATF7IP的表达减少了50%,进一步证明了乳腺癌相关的增强子是ATF7IP表达的调控子。通过scRNA-Seq鉴定sgRNA和整体mRNA丰度,所有表达特定sgRNA的细胞聚集在一起,并计算了sgRNA对顺式基因表达的影响。利用CRISPRqtl定义了16个风险基因座的靶基因,包含来自功能CRISPR筛选的INQUISIT-1预测到的18个基因。此外,研究者还研究了100个sgRNAS靶向50个基因的TSS。与以往报道一致,TSS靶向sgRNA对其靶基因的表达有显著的影响(p<0.0001),证明了该研究方法的可靠性。通过CRISPRqtl确定了chr19(19,048,246-20,048,246)增强子上的28个基因靶点,这很可能是由于分子多效性导致的。与功能筛选、HiChIP和荧光素酶实验结果一致,通过CRISPRqtl发现乳腺癌风险位点(靶向增强子)与ATF7IP之间存在强相互作用,证明使用多种检测方法定义GWAS靶点的价值。总计,通过表型CRISPR筛选并基于INQUISIT level 1方法下得分的21个基因中,发现与HiChIP中显示染色质相互作用的18个基因重叠,与CRISPRqtl鉴定的受这些增强子调控的10个基因重叠。
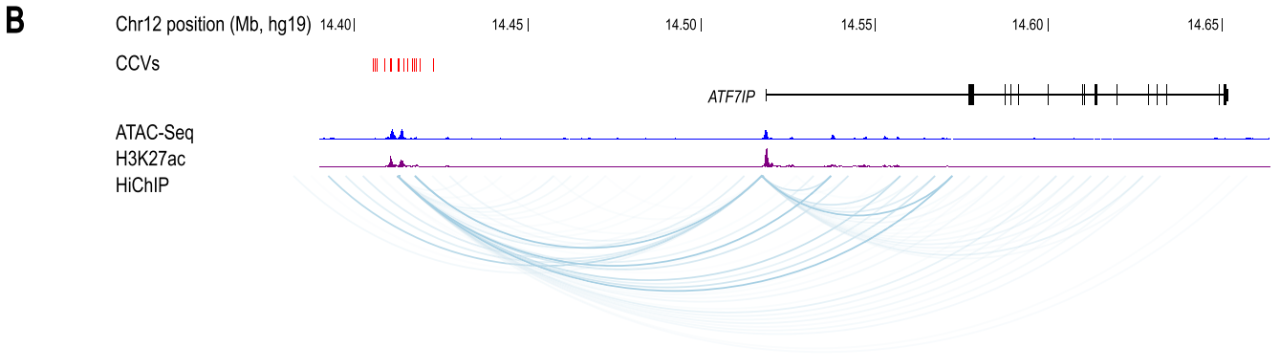
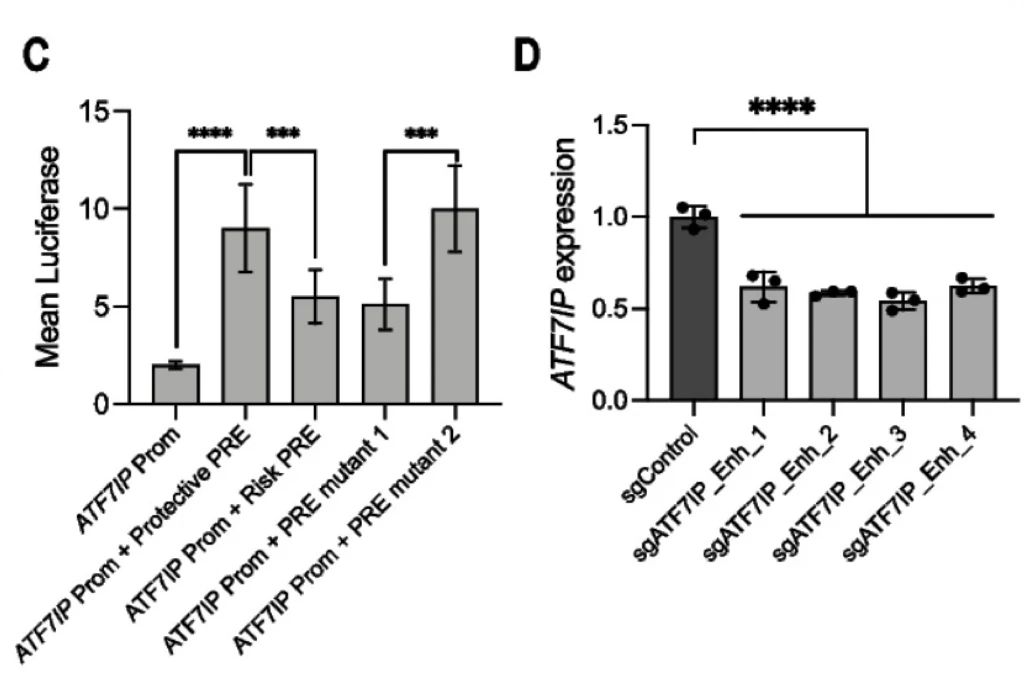
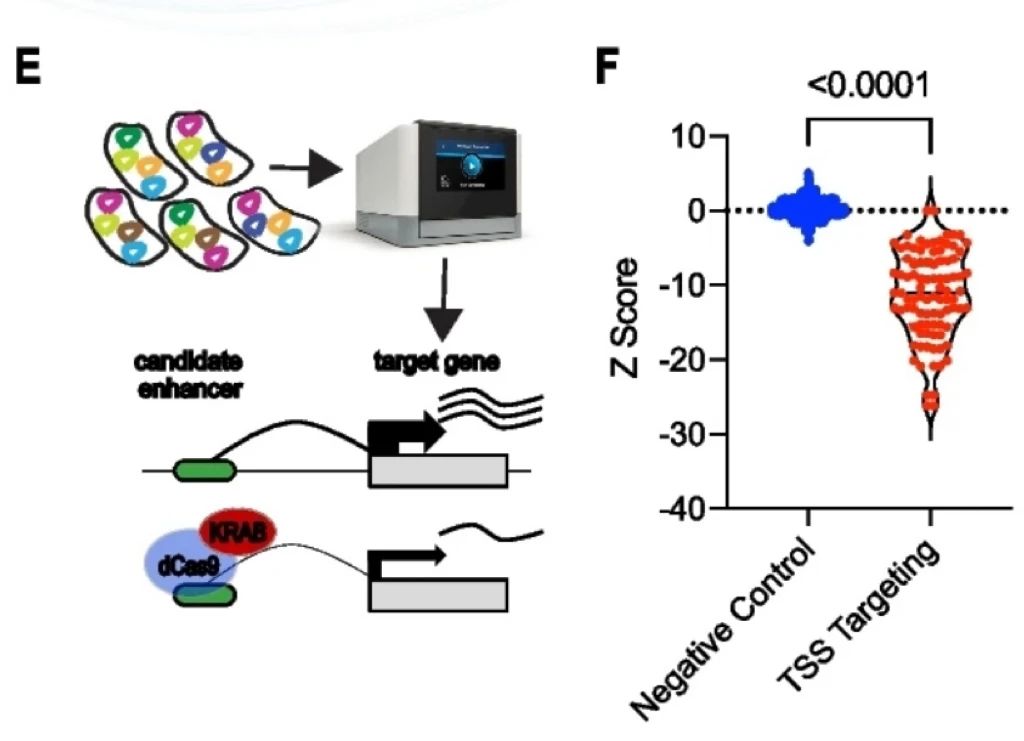
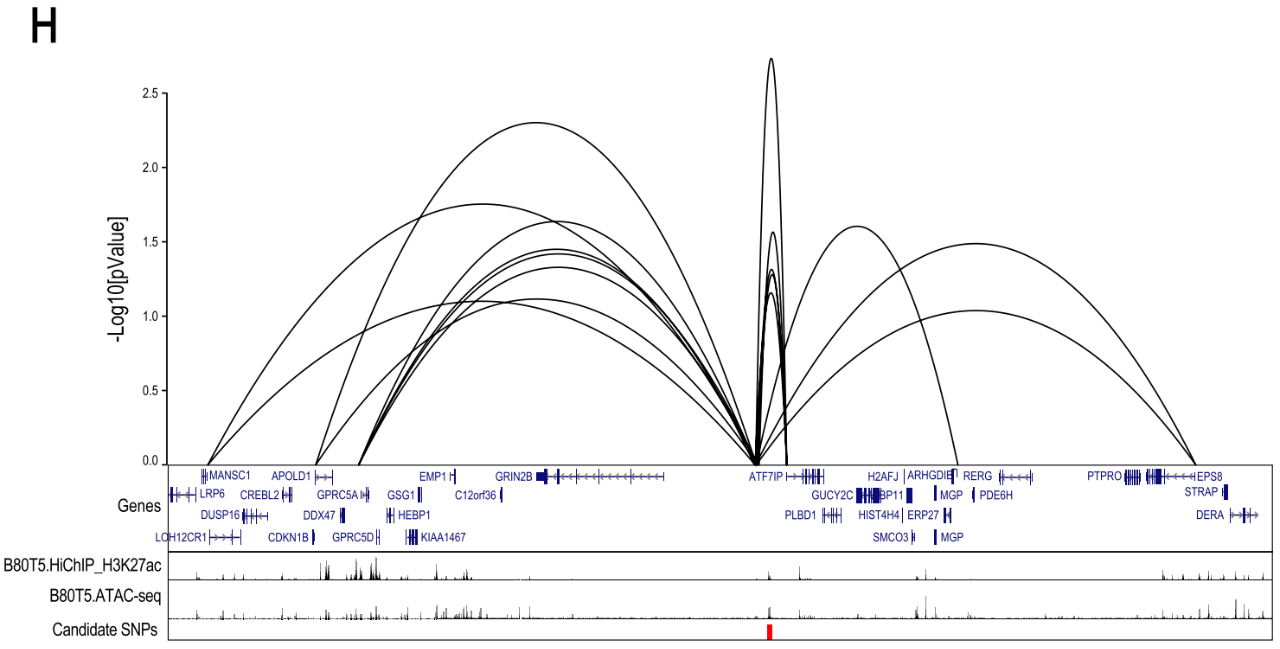

图4 染色体构象捕获技术和CRISPRqtl 证明乳腺癌风险基因座与功能筛选得分基因之间的相互作用
研究结论
研究表明功能性CRISPR筛选是一种用于识别与乳腺癌风险基因座相关基因的的可靠的且成本效益高的一种高通量方法。这种方法的应用和扩展对于利用癌症 GWAS数据的优势非常重要,并且在基因组研究转化治疗方面也发挥了重大作用。
表观生物HiChIP 产品介绍
HiChIP (in situ Hi-C followed by chromatin immunoprecipitation) 是一种利用原位Hi-C原理和转座酶介导构建文库来解析染色质构象的方法。该方法由Howard Chang实验室建立,相关方法论文在2016年11月份发表于 Nature Methods[4]。
HiChIP结合了Hi-C技术和ChIA-PET技术,用更小的数据量获取更高分辨率的染色质三维结构信息。通过技术革新,表观生物在国内首次推出最新的HiChIP技术服务!在三维构象水平研究DNA和蛋白接触点,挖掘平面测序无法揭示的相互作用!
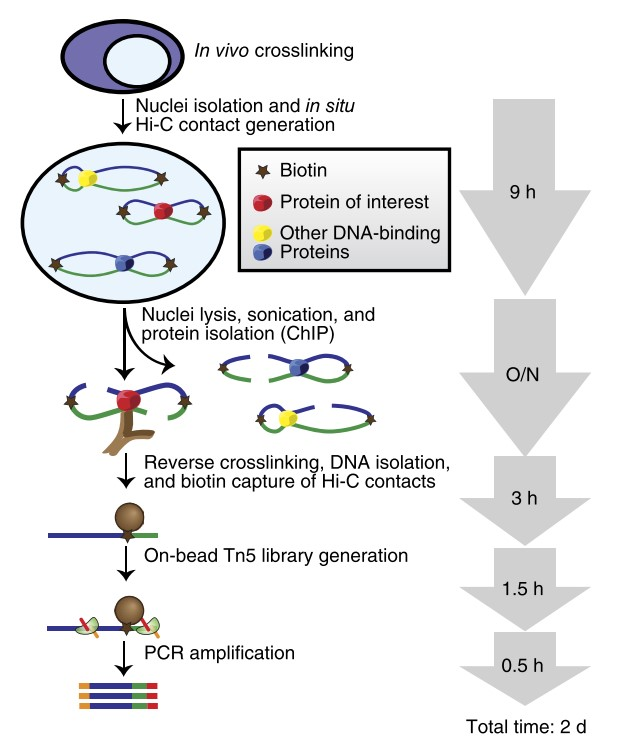
HiChIP通过裂解细胞前就在细胞核中交联,从而降低假阳性,最大化提高DNA contact的捕获效率。然后收集细胞核,在原位产生Hi-C交联,利用生物素标记DNA末端。接着进行细胞核的裂解,超声打断DNA,后续用特异性的抗体进行ChIP实验。得到DNA蛋白复合物后,进行DNA洗脱和反向交联。随后,进行生物素捕获Hi-C交联和文库制备,上机测序。
技术应用
1.针对特定蛋白的染色质互作构象分析2.转录因子作用机制研究3.表观修饰对基因调控的机制研究
技术优势
1. 构象信息读取的产量提高了10倍以上2. 相对于ChIA-PET,样本要求降低了100倍以上3. 所需细胞数量低于Hi-C4. 比Hi-C具有更大的信噪比
送样要求
细胞数量:1*10^7交联细胞,不低于5*10^6交联细胞;
测序方案
测序平台:NovaSeq 6000测序模式:PE150测序数据量:100G base(至少大于90G base)
生物信息分析内容
1.序列比对;
2.HiChIP实验质控分析(aligment、valid_interation_pairs);
3.HiChIP互作热图(多种分辨率);
4.Loop鉴定;
5.Loop注释(enhancer-promoter);
6.产生可视化文件;
7.Loop差异分析;
8.GO与KEGG分析;
9.Hub super-enhancer鉴定(可选分析);
10.高级分析(定制分析);
参考文献
1. Tuano, N.K., et al., CRISPR screens identify gene targets at breast cancer risk loci. Genome Biology, 2023. 24(1): p. 1-23.
2. Fachal, L., et al., Fine-mapping of 150 breast cancer risk regions identifies 191 likely target genes. Nature genetics, 2020. 52(1): p. 56-73.
3. Michailidou, K., et al., Association analysis identifies 65 new breast cancer risk loci. Nature, 2017. 551(7678): p. 92-94.
4. Mumbach, M.R., et al., HiChIP: efficient and sensitive analysis of protein-directed genome architecture. Nature methods, 2016. 13(11): p. 919-922.