新品上市 | Polysome-seq翻译组测序,评估翻译效率的“黄金标准”
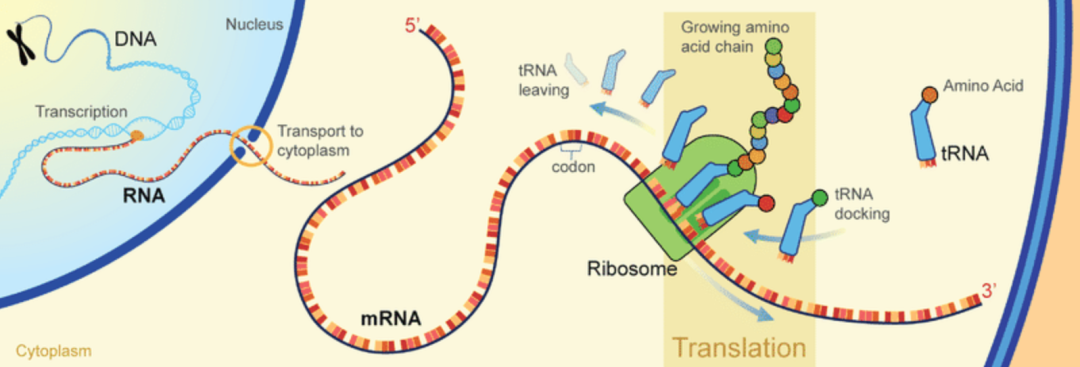
项目简介
翻译组是蛋白组的最佳体现者,是连接转录组和蛋白组的桥梁。Polysome profling是最经典的翻译组检测技术[1],该技术利用核糖体沉降系数较大的特性分离多聚核糖体,是评估翻译效率的“黄金标准”。
表观生物推出Polysome-seq技术服务,采用Biocamp全自动密度梯度制备与分析仪器,通过蔗糖密度梯度分离多聚核糖体,并绘制多聚核糖体峰图,结合高通量测序鉴定翻译效率差异的基因,助力从mRNA到蛋白质之间翻译水平调控机制研究。
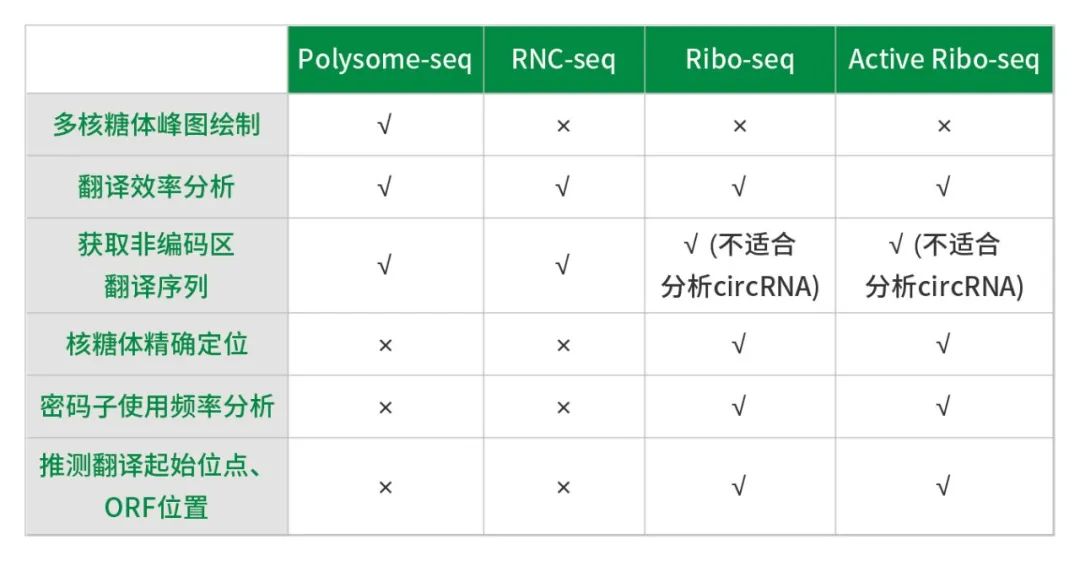
表1. 多种翻译组技术对比
应用与优势
技术应用(联合longRNA-seq)
1. 确定蛋白翻译效率
2. 研究翻译调控和基因表达情况,如目标mRNA降解、翻译抑制
3. 探究表观修饰的调控机制
4. 鉴定新生肽的共翻译降解
5. 研究翻译事件,如核糖体合成、组装、翻译相关因子
技术优势
1. 蔗糖密度梯度离心分离多聚核糖体,翻译效率“黄金标准”
2. 直观地反映细胞和组织内翻译的状态
3. 可以鉴定同一mRNA上核糖体数量
4. 可以检测非编码区、翻译调控信息
5. 适用于多种类型的样本

技术流程
送样要求
细胞,≥ 1×107 个细胞 / 样本
样本类型
≥ 100mg/ 样本
组织
仅限人、大小鼠,更低样本量及其他物种请咨询评估
样品物种:
分析内容
Polysome profiling
1. 原始数据过滤与测序质量评估
2. rRNA、tRNA 去除,reads 长度过滤
3. 参考基因组比对、参考转录组比对
4. 多聚核糖体峰图绘制
5. 全局翻译活性差异分析(Polysomes/Monosomes)
6. 差异翻译基因分析
7. 差异翻译基因热图(仅限有生物学重复)
8. 差异翻译基因GO、KEGG分析
9. 基因翻译效率计算(需要用 mRNA-seq 或 longRNA-seq)
10. 差异翻译效率分析
11. 差异翻译效率基因GO、KEGG分析
对照 RNA-seq 或 longRNA-seq 分析
1. 测序原始reads去接头,质量 控制
2. 参考基因组比对
3. 基因表达定量分析
4. 基因注释
5. 差异表达分析
6. 差异基因热图绘制(仅限有生物学重复)
8. 差异基因GO、KEGG分析
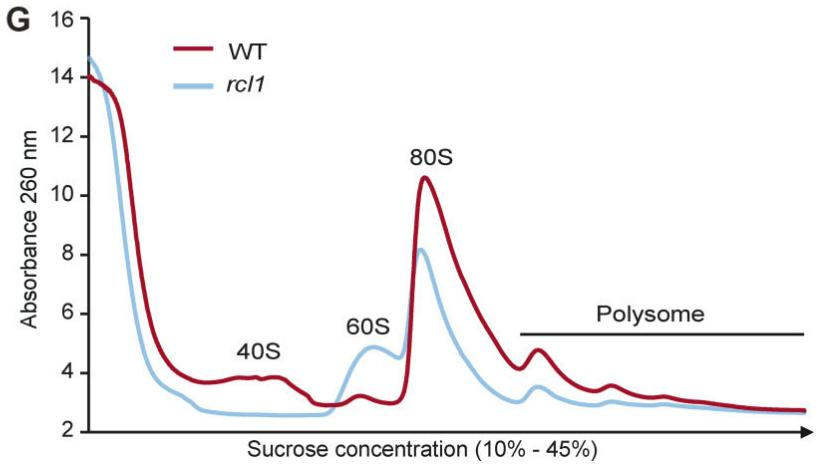
图1. 多聚核糖体峰图[2]
表观生物实测数据
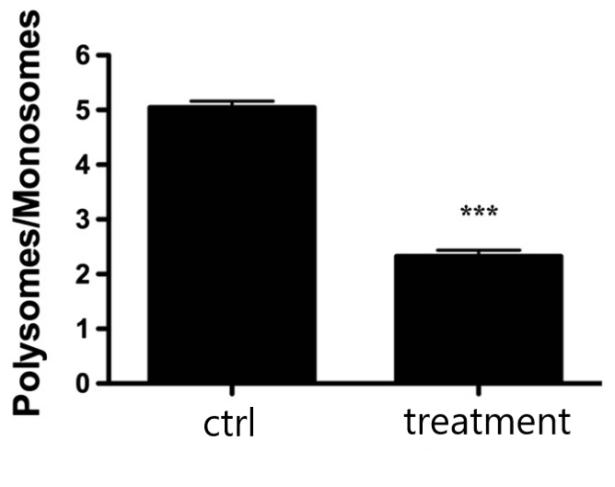
图2. 全局翻译活性分析[2]
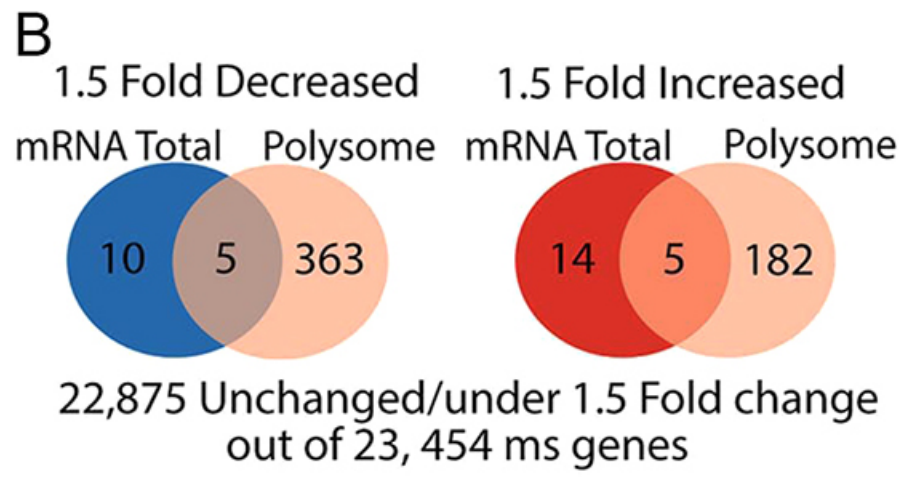
图2. 韦恩图对比总RNA和多聚核糖体丰度[3]
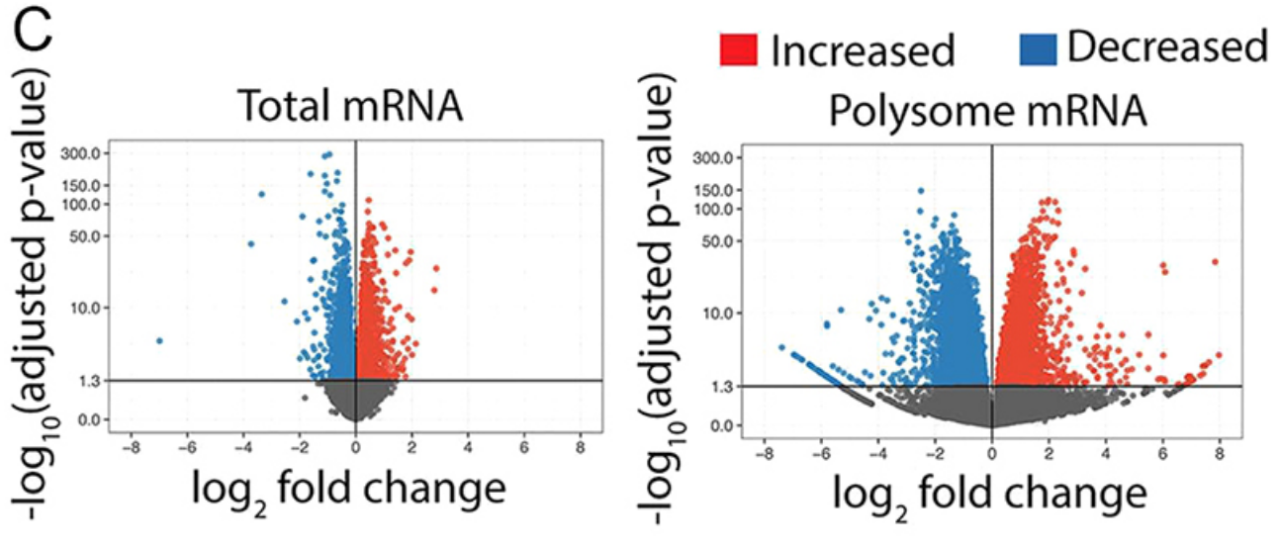
图3. 火山图分析差异翻译基因[3]
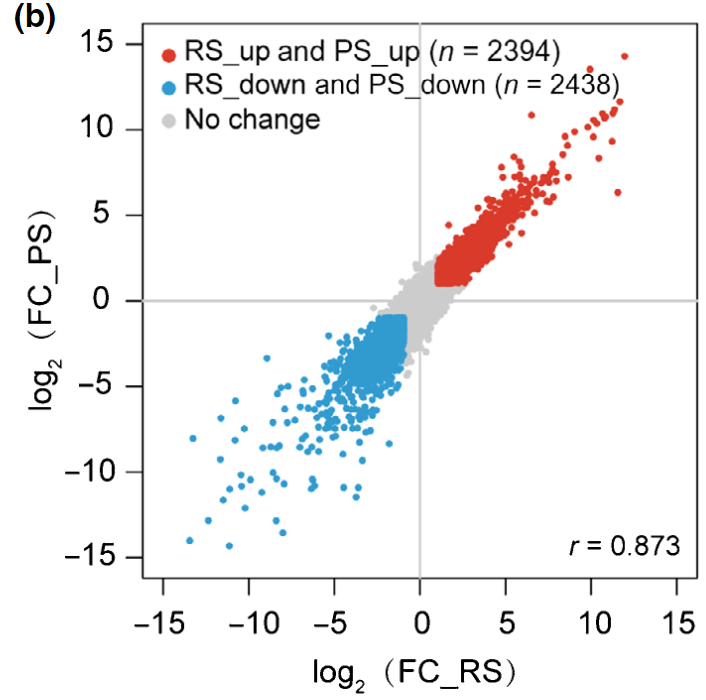
图4. 四象限图分析差异翻译基因。RS表示总RNA,PS表示多聚核糖体[4]
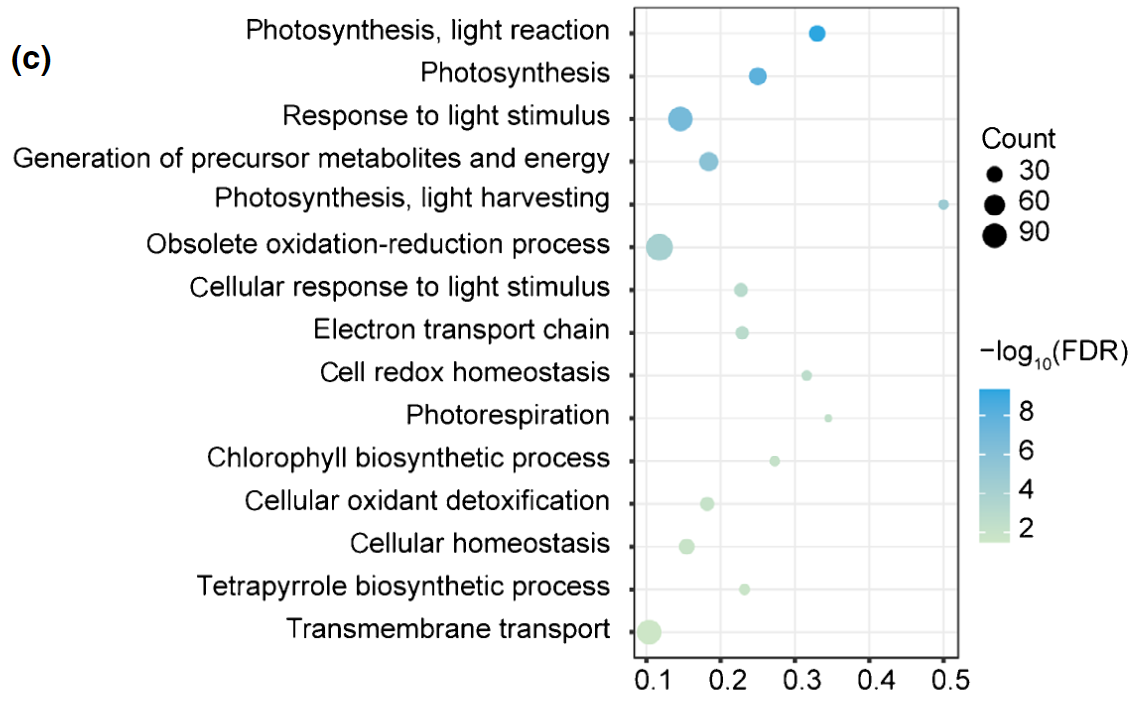
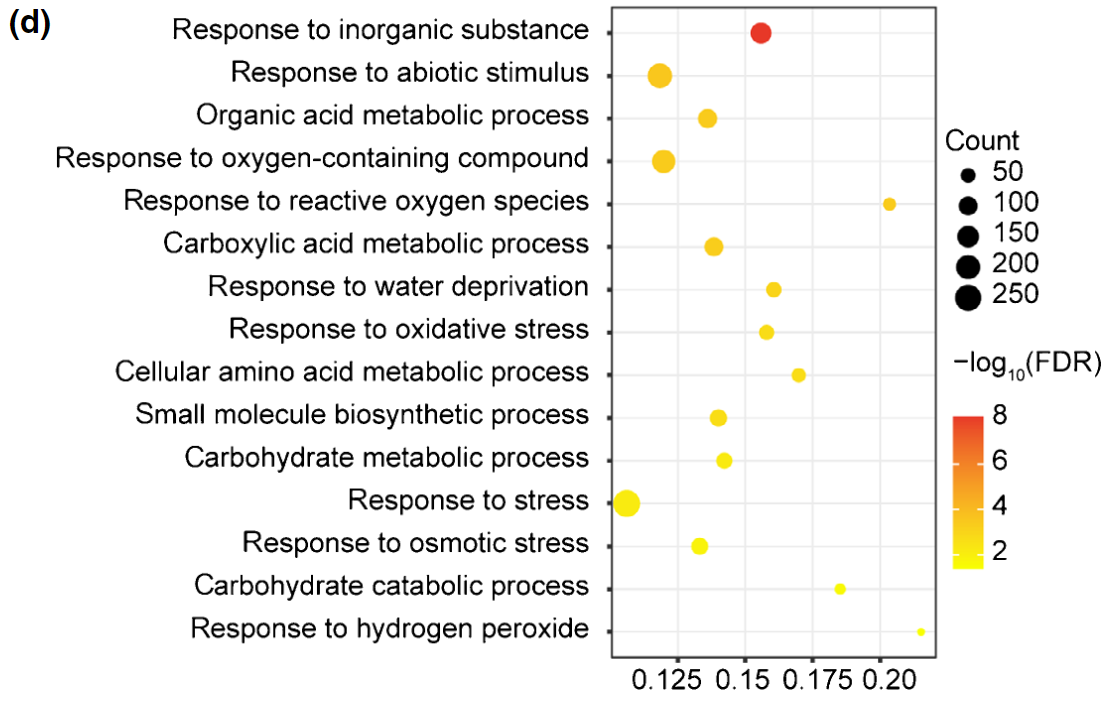
图5. 差异翻译基因GO分析[4]
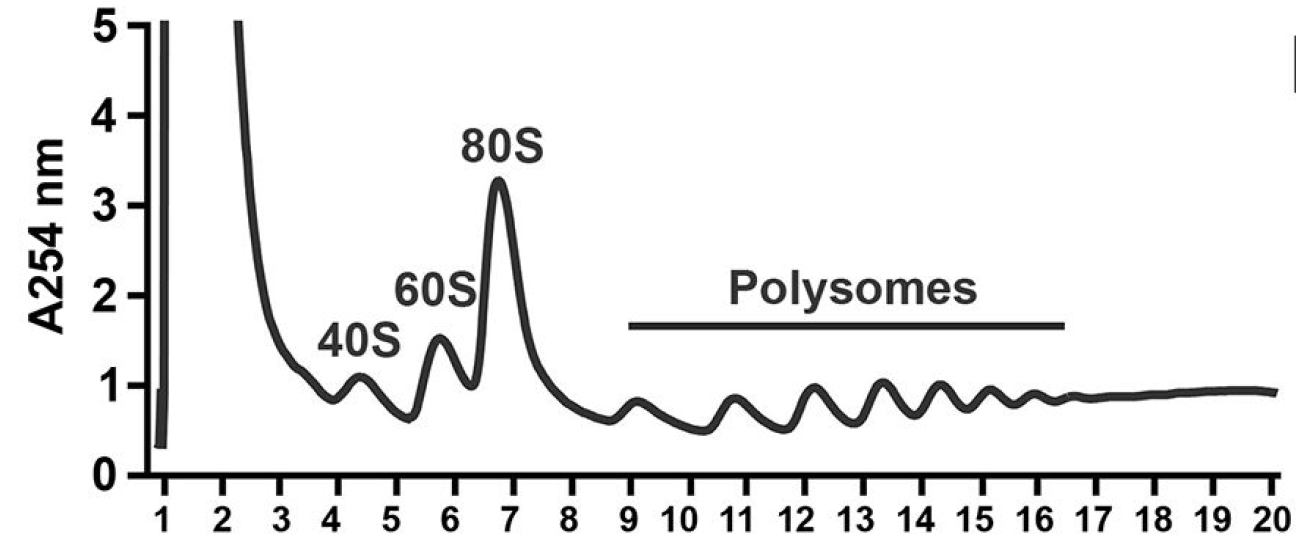
图6. 多聚核糖体峰图
案例分析
Nat Cell Biol:METTL16促进翻译和肿瘤发生且不依赖于m6A修饰[5]
此研究首次发现了METTL16的非m6A依赖作用,通过与eIF3a/b和rRNA的直接相互作用促进翻译的启动,证实了METTL16在肝癌中的促癌作用。
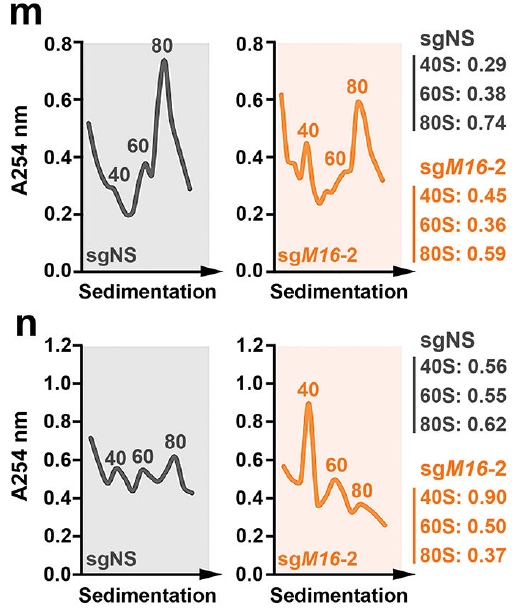
图7. 多聚核糖体峰图显示敲降METTL16后,80S核糖体减少
![Cell:QKI将m7G修饰转录本转运至应激颗粒并调节mRNA代谢[6]](http://openapi.whaleng.com/file-server/ep/2024/01/0ff894adfdbc4a5bb0746959e8bec5c0.png)
图8. Polysome profiling结果显示QK17过表达或删除对翻译效率无显著影响
Cell:QKI将m7G修饰转录本转运至应激颗粒并调节mRNA代谢[6]
此研究筛选并鉴定出mRNA内部m7G修饰的首个识别蛋白QKI (Quaking),而且报道了在应激状态下QKI对细胞应激颗粒内具有m7G修饰mRNA的动态调控作用。研究者通过高通量测序综合分析了应激状态下m7G meRIP-seq、QKI7 RIP-seq以及SG RNA-seq的数据,结果显示Quaking蛋白(QKIs)优先识别内部mRNA m7G修饰。接下来研究者开展了mRNA稳定性检测和Polysome profiling,结果表明在应激状态下QKI可以调控Hippo信号通路关键基因等靶基因的稳定性和翻译效率。
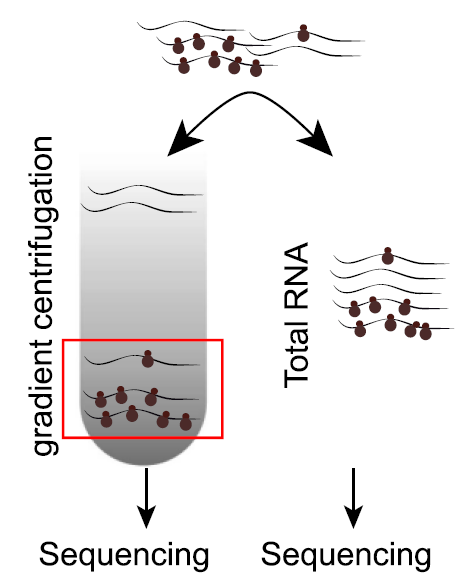
图9. Polysome-seq分析示意图
此研究证实了METTL1和WDR4在食管鳞癌(ESCC)中显著上调,并与ESCC预后不良相关。从机制上,研究者证明了tRNA m7G修饰通过调控m7G相关密码子富集mRNA的翻译,包括mTOR和自噬通路基因的负调控因子,为ESCC治疗靶向药物的开发提供线索。对METTL1敲降细胞以及对照细胞采用Polysome-seq计算了mRNA上的m7G tRNA所对应的密码子的数量。结果显示,mRNA上越多,翻译效率(TE)降低的mRNA具有明显更多的由m7G tRNA所对应的密码子。
Nat Commun:tRNA m7G修饰通过RPTOR/ULK1/自噬轴促进食管鳞癌发展
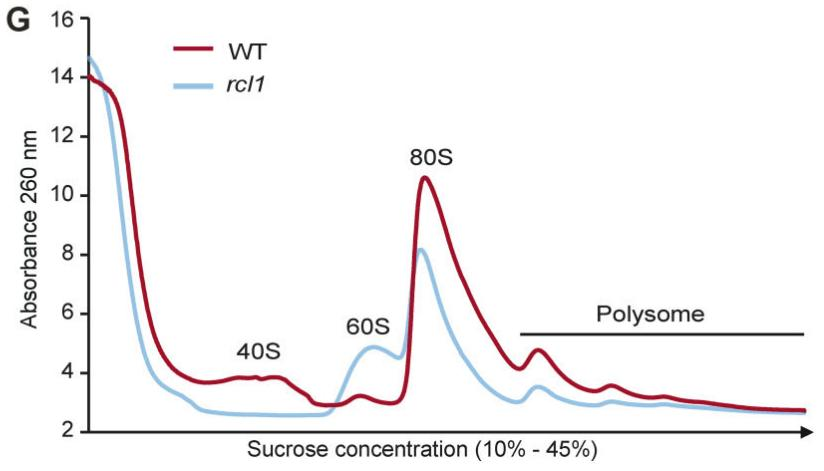
图10. 多聚核糖体峰图
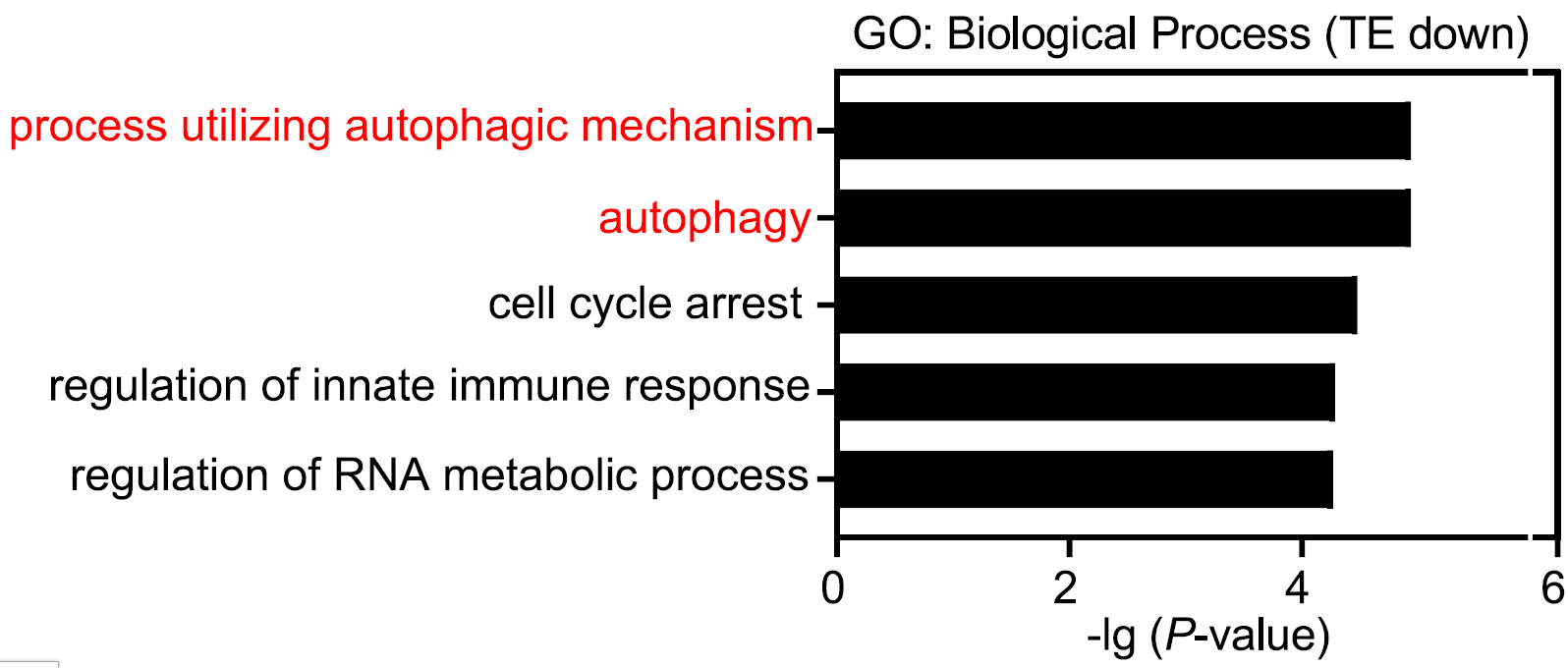
图11. 差异翻译效率基因GO分析
参考文献
[1] Kudla M, Karginov FV. 2016. Measuring mRNA Translation by Polysome Profiling. Methods Mol Biol 1421: 127-135.
[2] Wang T, Chang Y, Zhao K, et al. Maize RNA 3'-terminal phosphate cyclase-like protein promotes 18S pre-rRNA cleavage and is important for kernel development. Plant Cell. 2022 Apr 26;34(5):1957-1979.
[3] Herrlinger S, Shao Q, Yang M, et al. Lin28-mediated temporal promotion of protein synthesis is crucial for neural progenitor cell maintenance and brain development in mice. Development. 2019 May 28;146(10):dev173765.
[4] Wang Z, Sun J, Zu , et al. Pseudouridylation of chloroplast ribosomal RNA contributes to low temperature acclimation in rice. New Phytol 2022 Dec;236(5).
[5] Su R, Dong L, Li Y, et al. METTL16 exerts an m6A-independent function to facilitate translation and tumorigenesis. Nat Cell Biol. 2022 Feb;24(2):205-216.
[6] Zhao Z, Qing Y, Dong L, et al. QKI shuttles internal m7G-modified transcripts into stress granules and modulates mRNA metabolism. Cell. 2023 Jul 20;186(15):3208-3226.e27.
[7] Han H, Yang C, Ma J, et al. N7-methylguanosine tRNA modification promotes esophageal squamous cell carcinoma tumorigenesis via the RPTOR/ULK1/autophagy axis. Nat Commun. 2022 Mar 18;13(1):1478.
