单细胞转录组测序服务,开启单细胞探索之旅

达普Galaxy星海单细胞建库系统
项目简介
单细胞转录组测序是在单个细胞水平上对转录组进行高通量测序分析的一项技术,本质是对极微量核酸进行测序检测。
表观生物推出基于华大智造DNBelab C-TaiM4单细胞液滴生成仪的转录组测序服务,该设备基于液滴微流控原理,用于细胞的分选和标记,具有细胞捕获率高,建库测序成本低等优点,助力肿瘤、干细胞、发育等方向疾病机制研究、细胞特异性分析,发现标志物,开发药物等研究。

技术原理
DNBelab C-TaiM 4 基于Drop-seq的液滴捕获原理,将带有Cell Barcode的凝胶微珠(大磁珠)、带有oligo测序标签的凝胶微珠(小磁珠)和单个细胞包裹在同一个“油包水”液滴中。然后在液滴中将细胞裂解,单个细胞释放出的mRNA被大磁珠微珠的oligo-dT捕获,同时与小磁珠测序接头结合,随后在液滴中共同进行进行逆转录反应,破乳回收cDNA中间产物和Oligo产物后分别进行cDNA文库和Oligo文库的构建,最后上机测序,进行数据分析[1]。
技术优势
1. 配备独立4通道微流控通路,可灵活完成1~4个样本的细胞分离、标记
2. 细胞捕获率高,适用于较多样本类型
3. 建库测序成本更低,有效数据更多
送样要求
细胞或组织样本,具体要求请详询
样本类型
仅限人和大小鼠
样本物种
分析内容
基本分析
1.细胞tag处理
2.序列比对
3.UMI计数
4.低质量细胞过滤
5.主成分分析
6.无监督聚类分析
7.细胞亚群再分群分析
8.差异分析
高级分析
1.细胞类型注释
2.细胞间相互作用分析
3.细胞内基因调控网络分析
4.细胞分化轨迹分析
5.KEGG分析
6.GO分析

图1. Barcode Rank 统计图
结果示例

图2. 细胞聚类分析图

图3. 细胞类型注释
案例解读
Cancer Cell:绘制肾癌瘤内和相关区域的单细胞转录组和空间转录组图谱[2]
此研究绘制了12名肾脏肿瘤患者约27万个细胞的单细胞转录组图谱,然后用空间转录组学进行验证。单细胞转录组识别的新突变,有助于广泛地推断基质细胞的克隆性和谱系并追踪髓系细胞的分化。此外,研究发现了肿瘤细胞具有六个元程序以及上皮-间质转化程序在肿瘤-正常界面高度富集,并与IL1B+巨噬细胞共同定位,这为肾癌的治疗提供一个潜在的治疗靶点。
样本设置:
(1) 从12名肾癌患者的外周血、正常肾脏、肿瘤核心的四个不同的区域和肿瘤正常界面中采集组织,以及肾周脂肪、正常肾上腺、肾上腺转移和肿瘤血栓的组织,进行单细胞转录组测序
(2) 8名患者的11个肿瘤正常界面和5个肿瘤核心组织切片,进行了空间转录组学分析。
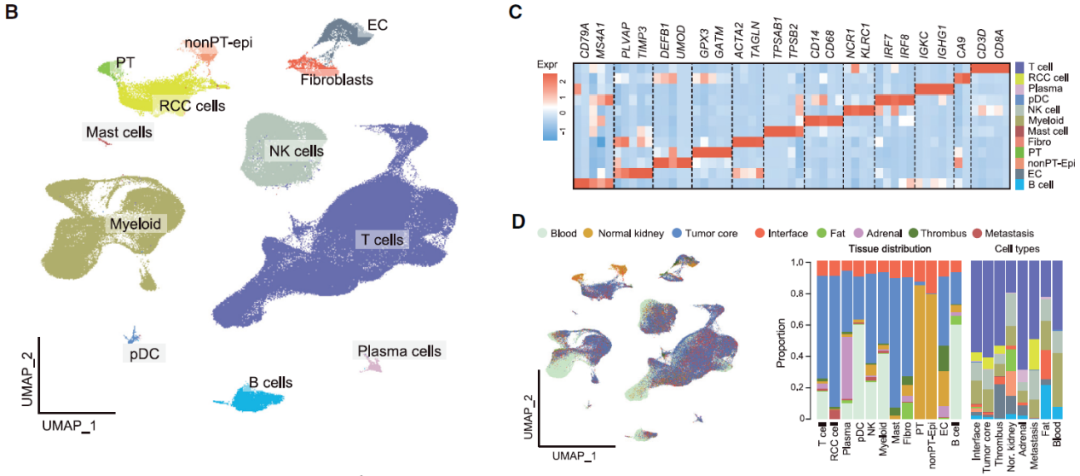
图7. 单细胞转录组测序结果,根据典型标记基因的表达,可将其大致分为12种主要细胞类型(图B、C),且并观察到不同的组织中具有不同细胞类型分布(图D)
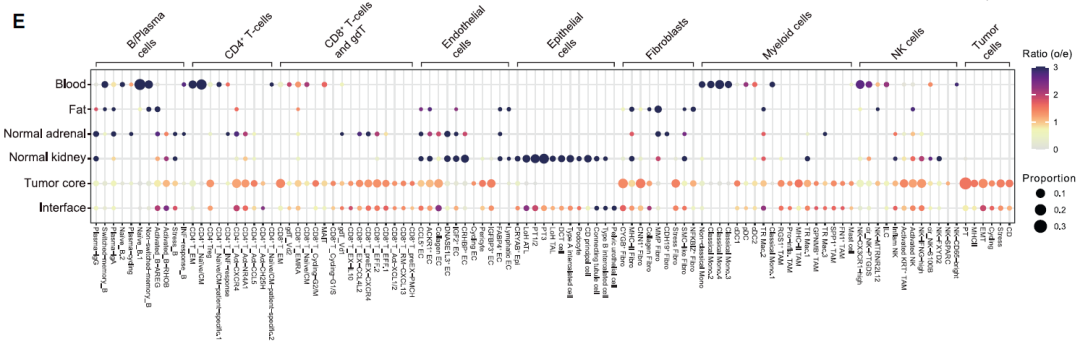
图8. 细胞亚群分析,确定不同组织中105个细胞亚群的差异性分布
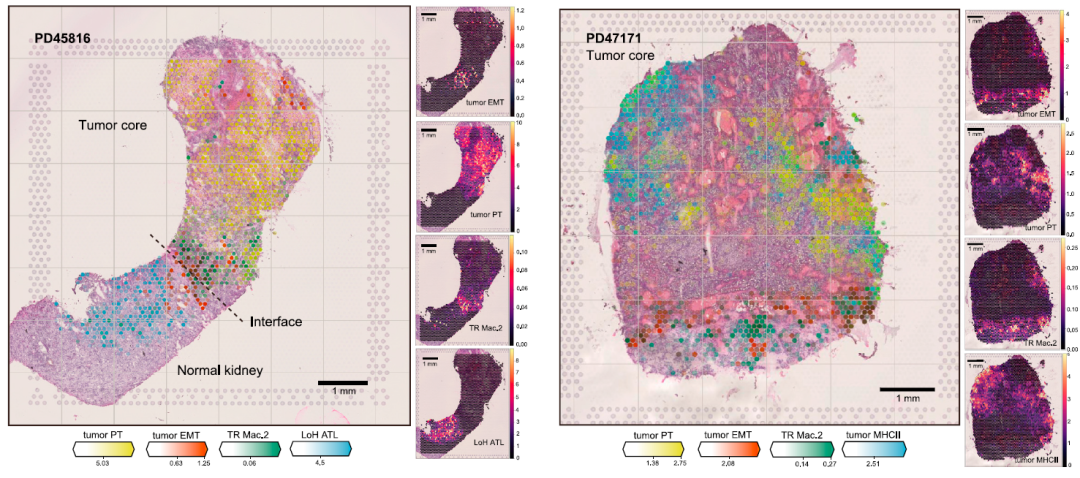
图9. 通过空间转录组分析,发现在多个组织切片中IL1B+巨噬细胞与EMThigh 肿瘤细胞的相关性最强
参考文献
[1] Jovic D, Liang X, Zeng H, Lin L, Xu F, Luo Y. Single-cell RNA sequencing technologies and applications: A brief overview. Clin Transl Med. 2022;12(3):e694. doi:10.1002/ctm2.694
[2] Li R, Ferdinand JR, Loudon KW, et al. Mapping single-cell transcriptomes in the intra-tumoral and associated territories of kidney cancer. Cancer Cell. 2022 Dec 12;40(12):1583-1599.e10.
[3] Zhang, S., Fang, W., Zhou, S. et al. Single cell transcriptomic analyses implicate an immunosuppressive tumor microenvironment in pancreatic cancer liver metastasis. Nat Commun 14, 5123 (2023).
[4] Cohen YC, Zada M, Wang SY, et al. Identification of resistance pathways and therapeutic targets in relapsed multiple myeloma patients through single-cell sequencing. Nat Med. 2021 Mar;27(3):491-503.
