RNA甲基化修饰m6Am-Exo-seq测序技术服务

项目简介
RNA甲基化修饰m6Am-Exo-seq
RNA上存在一百多种化学修饰,其中N6-甲基腺嘌呤(m6A)作为真核生物mRNA上含量最普遍的化学修饰,参与调控了很多重要的生物学过程。不同于m6A,m6Am主要位于真核生物mRNA 5'端帽子之后的第一个碱基。早在1975年,科学家就鉴定到了m6Am的存在。m6Am也是一个动态、可逆的修饰,但其修饰酶PCIF1近两年才发现。m6Am修饰主要位于5’m7Gppp帽子后的第一个碱基,主要通过影响mRNA的蛋白翻译效率发挥作用。
为推动表观转录修饰m6Am的研究,表观生物最新开发m6Am-Exo-seq测序技术,大幅降低样本要求量,可以在全转录组范围(mRNA及lincRNA)高效准确的鉴定m6Am的修饰位点及丰度变化。
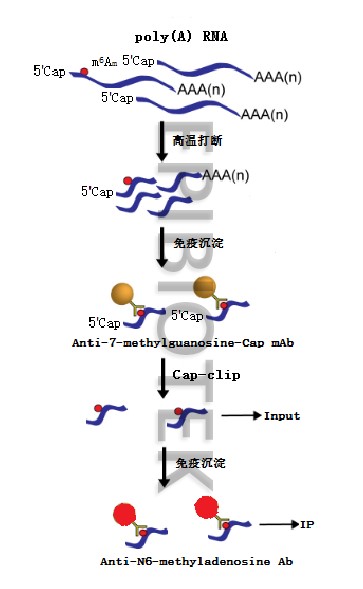
项目流程
送样要求
100 ug
总RNA
5x10⁷个
细胞数量
100 mg
组织
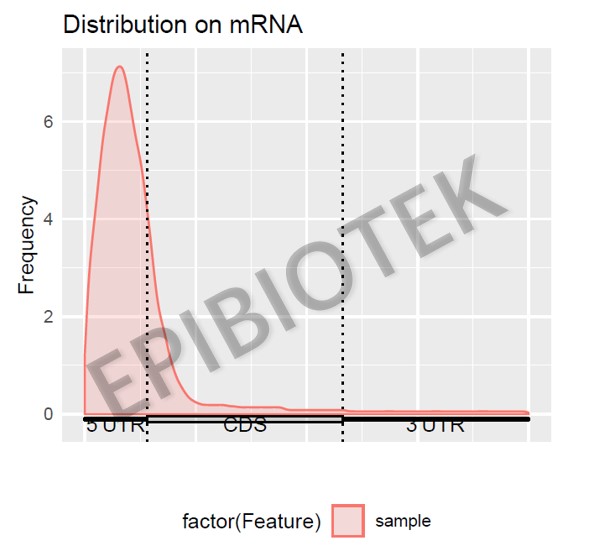
表观生物实测数据
Peak在mRNA结构上的分布metageneplot图
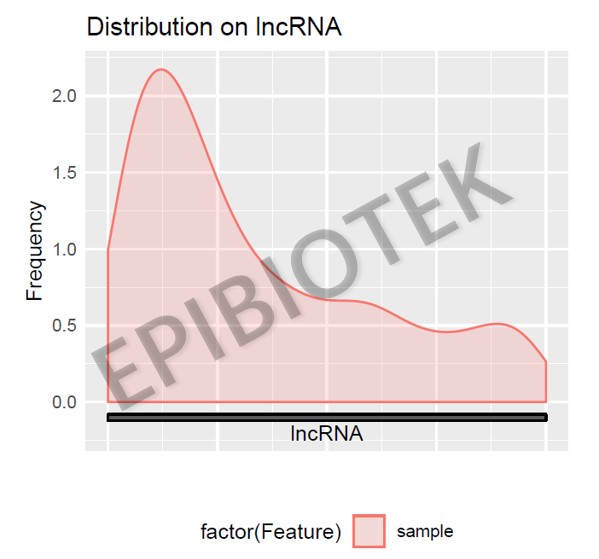
表观生物实测数据
Peak在lincRNA结构上的分布metageneplot图

motif分析结果(BCA特征,不同于m6A修饰的GRACH)
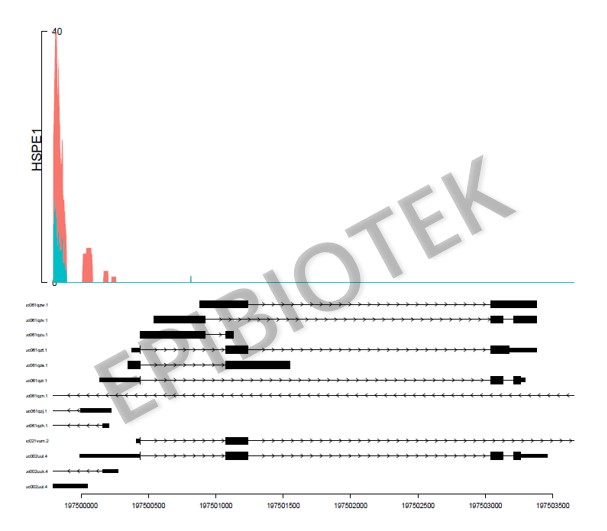
IGV特定基因峰图
基本分析
1.测序原始reads去接头,质量控制(QC)
2.参考基因组比对(Mapping)
3.富集区域鉴定(PeakCalling)
4.富集区域注释(PeakAnno)
5.lncRNA修饰分析、mRNA修饰分析
6.富集区域metagene plot图、pie plot图、venny图
7.Motif分析
8.Peak基因GO和KEGG分析
9.差异Peak分析(即差异RNA修饰mRNA、lncRNA)
10.差异Peak基因GO和KEGG分析
高级分析
1.单位点修饰预测分析(SingleSite)
2.差异RNA修饰热图(Heatmap)
3.累计分布曲线分析(Cumulative Distribution Fraction Analysis)
4.关联分析(与RNA-seq 关联分析或多组学关联分析)
5.关联分析四象限图
6.关联分析热图(Heatmap)
7.IGV峰图(附5个基因)
参考文献
1.Sendinc E, Valle-Garcia D, Dhall A, et al. PCIF1 catalyzes m6Am mRNA methylation to regulate gene expression[J]. Molecular cell, 2019, 75(3): 620-630. e9.
2.Mauer J, Sindelar M, Despic V, et al. FTO controls reversible m 6 Am RNA methylation during snRNA biogenesis[J]. Nature chemical biology, 2019, 15(4): 340-347.
3.Akichika S, Hirano S, Shichino Y, et al. Cap-specific terminal N6-methylation of RNA by an RNA polymerase II–associated methyltransferase[J]. Science, 2019, 363(6423): eaav0080.
4.Sun H, Zhang M, Li K, et al. Cap-specific, terminal N 6-methylation by a mammalian m 6 Am methyltransferase[J]. Cell research, 2019, 29(1): 80-82.
5.Li K, Cai J, Zhang M, et al. Landscape and regulation of m6A and m6Am methylome across human and mouse tissues[J]. Molecular cell, 2020, 77(2): 426-440. e6.
