[新品上线]RNA甲基化修饰 m6Am-Exo-seq测序技术服务隆重上市
项目简介:
RNA上存在一百多种化学修饰,其中N6-甲基腺嘌呤(m6A)作为真核生物mRNA上含量最普遍的化学修饰,参与调控了很多重要的生物学过程。不同于m6A,m6Am主要位于真核生物mRNA 5'端帽子之后的第一个碱基。早在1975年,科学家就鉴定到了m6Am的存在。m6Am也是一个动态、可逆的修饰,但其修饰酶PCIF1近两年才发现。m6Am修饰主要位于5’m7Gppp帽子后的第一个碱基,主要通过影响mRNA的蛋白翻译效率发挥作用。
为推动表观转录修饰m6Am的研究,表观生物最新开发m6Am-Exo-seq测序技术,大幅降低样本要求量,可以在全转录组范围(mRNA及lincRNA)高效准确的鉴定m6Am的修饰位点及丰度变化。
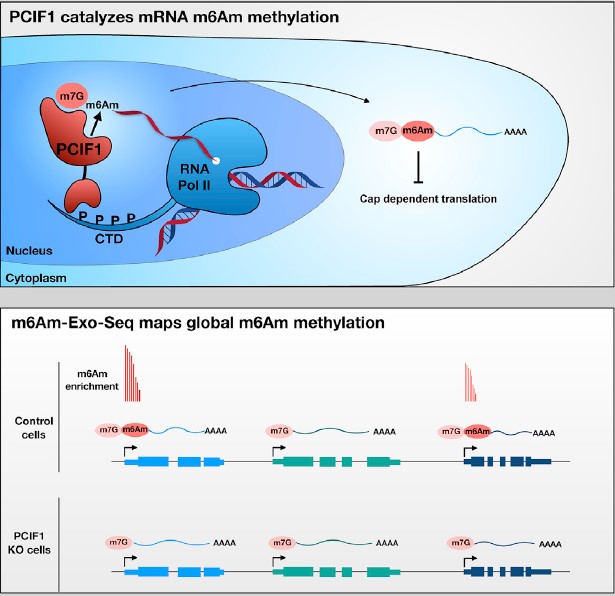
图1. m6Am修饰反应及作用机制[1]
技术流程:
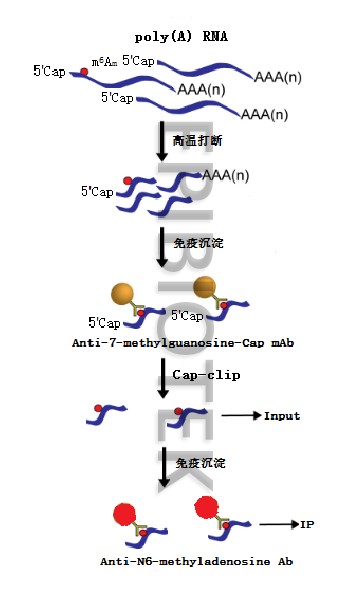
图2. m6Am-Exo-seq双IP建库流程
送样要求:
样本类型:
1. 总RNA,100ug
2. 细胞,数量为5x107
3. 组织,质量为100mg
样本物种:
仅限人、大小鼠物种,其他物种需评估
表观生物实测数据:
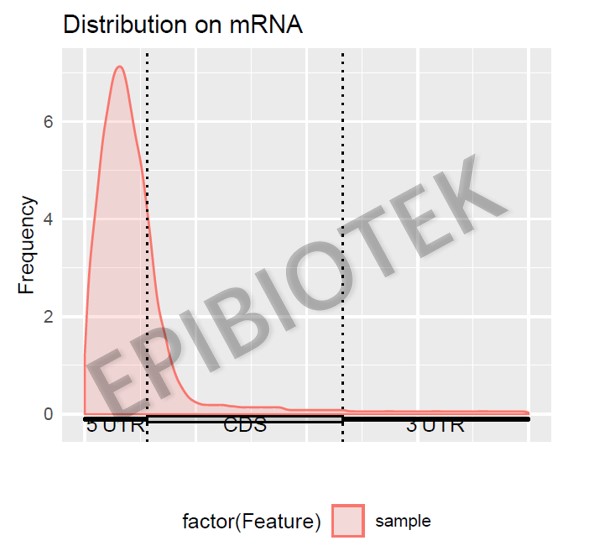
图3. Peak在mRNA结构上的分布metageneplot图
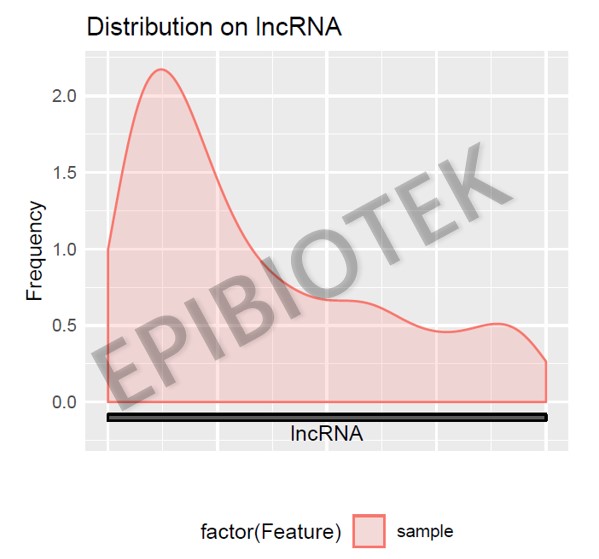
图4. Peak在lincRNA结构上的分布metageneplot图

图5. motif分析结果(BCA特征,不同于m6A修饰的GRACH)
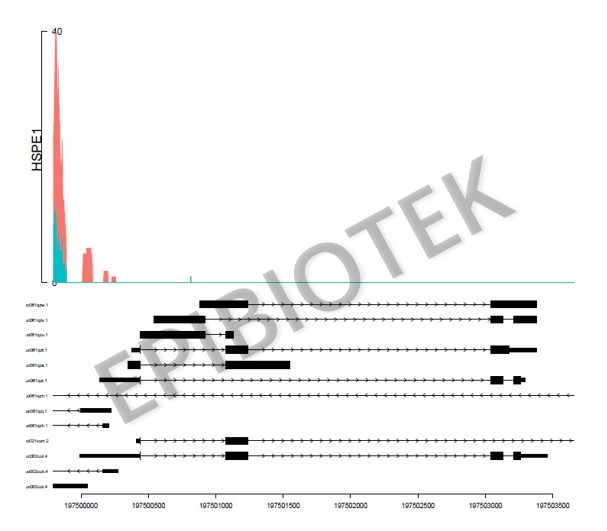
图6. IGV特定基因峰图
基本分析
1.测序原始reads去接头,质量控制(QC)
2.参考基因组比对(Mapping)
3.富集区域鉴定(PeakCalling)
4.富集区域注释(PeakAnno)
5.lncRNA修饰分析、mRNA修饰分析
6.富集区域metagene plot图、pie plot图、venny图
7.Motif分析
8.Peak基因GO和KEGG分析
9.差异Peak分析(即差异RNA修饰mRNA、lncRNA)
10.差异Peak基因GO和KEGG分析
高级分析
1.单位点修饰预测分析(SingleSite)
2.差异RNA修饰热图(Heatmap)
3.累计分布曲线分析(Cumulative Distribution Fraction Analysis)
4.关联分析(与RNA-seq 关联分析或多组学关联分析)
5.关联分析四象限图
6.关联分析热图(Heatmap)
7.IGV峰图(附5个基因)
参考案例:
1.Sendinc E, Valle-Garcia D, Dhall A, et al. PCIF1 catalyzes m6Am mRNA methylation to regulate gene expression[J]. Molecular cell, 2019, 75(3): 620-630. e9.
2.Mauer J, Sindelar M, Despic V, et al. FTO controls reversible m 6 Am RNA methylation during snRNA biogenesis[J]. Nature chemical biology, 2019, 15(4): 340-347.
3.Akichika S, Hirano S, Shichino Y, et al. Cap-specific terminal N6-methylation of RNA by an RNA polymerase II–associated methyltransferase[J]. Science, 2019, 363(6423): eaav0080.
4.Sun H, Zhang M, Li K, et al. Cap-specific, terminal N 6-methylation by a mammalian m 6 Am methyltransferase[J]. Cell research, 2019, 29(1): 80-82.
5.Li K, Cai J, Zhang M, et al. Landscape and regulation of m6A and m6Am methylome across human and mouse tissues[J]. Molecular cell, 2020, 77(2): 426-440. e6.
- - - 推荐阅读 - - -

杨建华教授线上讲座免费听!表观学院线上课堂第二弹重磅上线!!
从2018年开始,表观生物已陆续举办了两届医学表观遗传学前沿技术培训班,以及三期表观生物专题研习班,这5次现场学习学员人数逾600人!为让更多对表观
2020-05-07

暖春钜惠!m6A测序8折!免费赠送万元高级分析内容!
即日起至4月30日 4个样本起,可享9折优惠 8个样本起,可享8折优惠 并赠送万元高级分析内容! 项目简介: RNA甲基化指发生
2020-03-15

表观生物关于安全有序复工的通知
即日起,表观生物除生产部以外的其他部门全面开展线上办公模式。预计2月15日起,表观生物各员工将开始在公司现场办公。
2020-02-12
