Nature丨朱冰课题组发现旁着丝粒异染色质的建立机制
以组蛋白H3K9甲基化修饰为标记的旁着丝粒异染色质(pericentric heterochromatin)是染色质的重要组成部分,其在确保正确的有丝分裂、调控恰当的基因表达以及维持基因组稳定性等方面起着重要作用【1-3】。异染色质这一名词于1928年由德国科学家Emil Heitz提出,被用来描述细胞中被染料浓密着色的染色质区域【4】,这类异染色质在旁着丝粒区尤为明显,并且在绝大部分细胞类型中均为异染色质,因此被称为组成型异染色质,以与由H3K27甲基化修饰所标记的兼性异染色质相区别。
组成型异染色质与1930年发现的首个表观遗传学现象(果蝇眼睛的位置效应花斑现象,position effect variegation,PEV)密切相关【5】。PEV现象的发现者Hermann Muller于1946年获得了诺贝尔奖。随后,科学家们通过遗传筛选获得了突变后能抑制该现象的基因,并将它们称为花斑效应抑制子基因Su(var)【6】。1986年, Su(var)2-5基因被克隆,其编码的蛋白质定位于组成型异染色质区域,所以被命名为异染色质蛋白1(heterochromatin protein 1,HP1)【7】。2000年,Su(var)3-9同源基因编码的SUV39H家族蛋白被发现是组蛋白H3K9甲基化酶【8】。该发现具有里程碑意义,因为SUV39H家族蛋白是首个组蛋白甲基化酶,H3K9甲基化也是首个能标记特定染色质区域的组蛋白甲基化修饰,这一工作开启了组蛋白甲基化修饰的研究浪潮。2001年,多个课题组发现HP1及其同源蛋白能够选择性识别甲基化的H3K9【9-11】。
然而,组成型异染色质领域仍然存在着两个长期的未解之谜:1.在高等真核生物中,SUV39H家族蛋白是怎样被招募到旁着丝粒区域以起始异染色质形成的?2.不同物种的旁着丝粒区域序列不保守,那么,为什么这种不保守的序列能被保守的分子机器所识别?
2024年7月4日,中国科学院生物物理研究所朱冰课题组在Nature发表题为Targeting pericentric non-consecutive motifs for heterochromatin initiation的研究论文【12】。该工作揭开了上述两个重要问题的谜底。

基于CRISPR/Cas9系统的特异性靶向能力以及APEX2系统的邻近标记能力,该研究首先开发了一种能够鉴定特定基因组位点附近蛋白质组的技术,并使用该技术鉴定到了能特异性地定位于旁着丝粒异染色质区域的锌指蛋白ZNF512和ZNF512B。
基于导入了外源性256×LacO序列的报告细胞系【13】,作者发现,当把锌指蛋白ZNF512和ZNF512B靶向到256×LacO序列上时,能在该重复序列上从头建立H3K9me3修饰和异染色质形态。在内源性的旁着丝粒区域,ZNF512和ZNF512B也能够招募SUV39H家族蛋白,并起始H3K9甲基化修饰的从头建立。这是因为ZNF512和ZNF512B具有与SUV39H家族蛋白直接相互作用的能力。
ZNF512和ZNF512B都有三个在脊椎动物中高度保守的锌指结构域,且其中负责与DNA发生碱基特异性识别的指纹氨基酸残基完全相同,这意味着每一个锌指能够结合DNA上相同的三个碱基(被预测和证实为TTC)。然而,无论是小鼠还是人的旁着丝粒区域都不存在由连续三个TTC构成的TTCTTCTTC序列,但不同物种的ZNF512和ZNF512B却能跨物种定位于不同物种来源的细胞的旁着丝粒区域,且该定位依赖于这些锌指结构域的指纹氨基酸残基。那么保守的DNA结合蛋白是如何识别不保守的靶DNA序列呢?
不同于将多个锌指结构域连续排布的经典锌指蛋白,ZNF512和ZNF512B的锌指结构域之间具有较长的连接序列,这暗示着它们可能结合不连续的TTC序列。为了验证这一假设,作者将锌指间较长的连接序列进行突变,将原本分离的锌指结构域排布成经典锌指蛋白样的连续锌指结构域模式,并发现此类突变后的蛋白质失去了异染色质定位能力,从而证实了ZNF512和ZNF512B在旁着丝粒异染色质上的定位依赖于其锌指结构域间较长的连接序列。
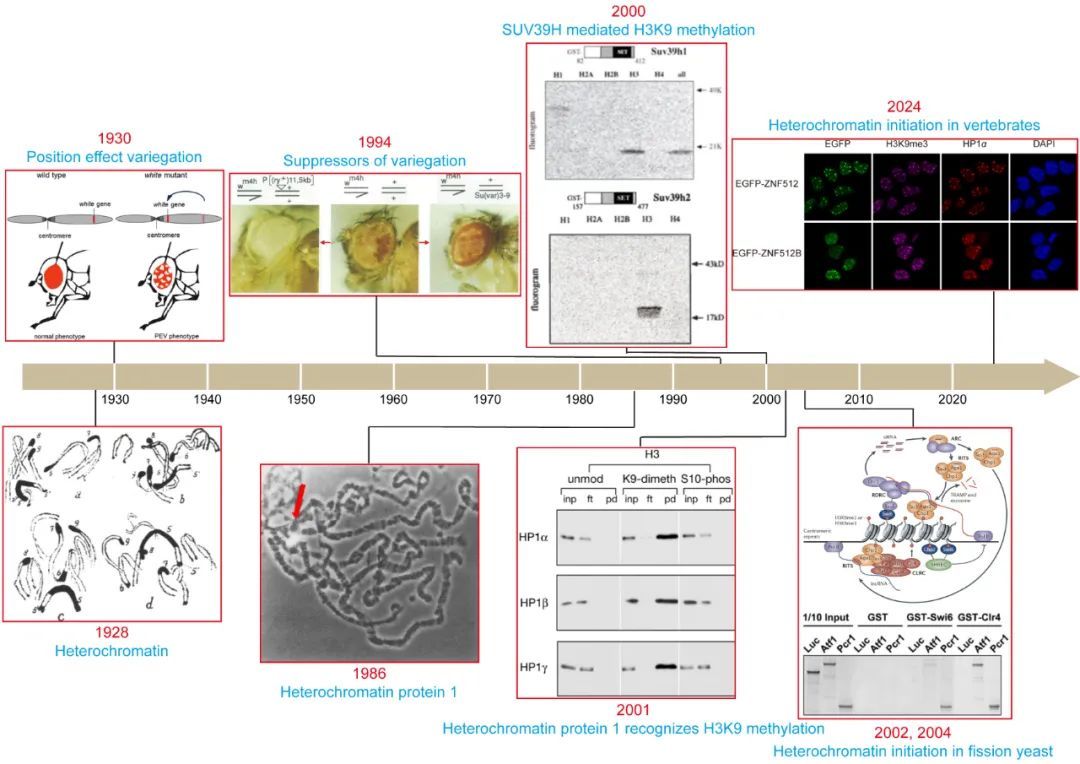 图:组成型异染色质的研究历史。
图:组成型异染色质的研究历史。
该论文的实验部分主要由马润泽博士完成,生物信息分析部分主要由张彦博士完成,他们为该论文的共同第一作者,张晶博士、张品奇博士、刘泽奇、樊一鸣在实验方面提供了帮助,王昊天博士和张珠强博士在生物信息分析部分做出了贡献。中国科学院生物物理研究所朱冰研究员为该论文通讯作者。感谢生物物理研究所/武汉大学的李国红研究员、北京生命科学研究所的杜立林研究员在实验过程中提供的宝贵建议,感谢索芳、张启祥、张浩楠、贾俊英、孟姝、王继峰、张朦朦的帮助。
原文链接:https://doi.org/10.1038/s41586-024-07640-5
参考文献
1. Allshire, R. C. & Madhani, H. D. Ten principles of heterochromatin formation and function. Nat. Rev. Mol. Cell Biol. (2018).
2. Janssen, A., Colmenares, S. U. & Karpen, G. H. Heterochromatin: guardian of the genome. Annu. Rev. Cell Dev. Biol. (2018).
3. Grewal, S. I. S. & Jia, S. Heterochromatin revisited. Nat. Rev. Genet. (2007).
4. Heitz, E. Das heterochromatin der moose (Bornträger, 1928).
5. Muller, H. J. Types of visible variations induced by x-rays in Drosophila. J. Genet. (1930).
6. Fodor, B. D., Shukeir, N., Reuter, G., Jenuwein, T. Mammalian Su(var) genes in chromatin control. Annu Rev Cell Dev Biol. (2010)
7. James, T. C., Elgin, S.C. Identification of a nonhistone chromosomal protein associated with heterochromatin in Drosophila melanogaster and its gene. Mol Cell Biol. (1986).
8. Rea, S. et al. Regulation of chromatin structure by site-specific histone H3 methyltransferases. Nature. (2000).
9. Lachner, M., O’Carroll, D., Rea, S., Mechtler, K. & Jenuwein, T. Methylation of histone H3 lysine 9 creates a binding site for HP1 proteins. Nature. (2001).
10. Nakayama, J., Rice, J. C., Strahl, B. D., Allis, C. D. & Grewal, S. I. S. Role of histone H3 lysine 9 methylation in epigenetic control of heterochromatin assembly. Science. (2001).
11. Bannister, A. J et al. Selective recognition of methylated lysine 9 on histone H3 by the HP1 chromo domain. Nature. (2001).
12. Ma, R. et al. Targeting pericentric non-consecutive motifs for heterochromatin initiation. Nature. (2024).13. Robinett, C. C. et al. In vivo localization of DNA sequences and visualization of large-scale chromatin organization using lac operator/repressor recognition. J. Cell Biol. (1996).