一文读懂超级增强子:从鉴定到跨组学深入机制研究
什么是超级增强子
说起超人,你一定知道;但是,超级增强子,你听过吗?作为目前国内还比较冷门的科研领域,他简直就是细胞中的超人!

图1. 近年来以超级增强子为主题的已发表文章数目
(IF≥10为高分文章,数据来源:Pubmed)
超级增强子(Super enhancer, SE)是一类具有超强转录激活特性的顺式调控元件,2013年由美国美国白头生物医学研究所(Whitehead Institute for Biomedical Research)学者Richard A. Young首次提出。与普通增强子(Typical enhancer, TE)相比,超级增强子区域跨度范围通常可达 8-20 Kb,远高于普通增强子的200-300 bp跨度范围。更重要的是,超级增强子比普通增强子具有更高密度的转录激活相关组蛋白修饰(H3K27ac、H3K4me1等)、Mediator复合体和Bromodomain containing 4 蛋白(BRD4,与组蛋白乙酰化修饰位点结合)和其他转录因子。
知识卡片:超级增强子的特征
以下特点决定了超级增强子具有强大的调控功能[1]:
(1) 超级增强子具有高密度的H3K27ac 和H3K4me1 修饰,结合有Mediator复合体和BRD4;
(2) 超级增强子结合的转录因子以及与转录活性相关的染色体的标记比普通增强子高很多;
(3) 超级增强子调控的基因比普通增强子调控的基因表达水平高很多
(4) 组成超级增强子的单个增强子也可以像普通增强子一样激活基因转录;
(5) 超级增强子可以结合组织中特异的转录因子;
(6) 与普通增强子相比,超级增强子活性对于转录因子的阻断更敏感。

图2. 超级增强子是具有转录活性增强子的一个大簇,富集高密度的关键转录因子、辅因子和增强子表观修饰标记[2]
超级增强子与疾病
目前,超级增强子已被报道与多种疾病发生发展有关,其中包括了不同类型的肿瘤(见下图):
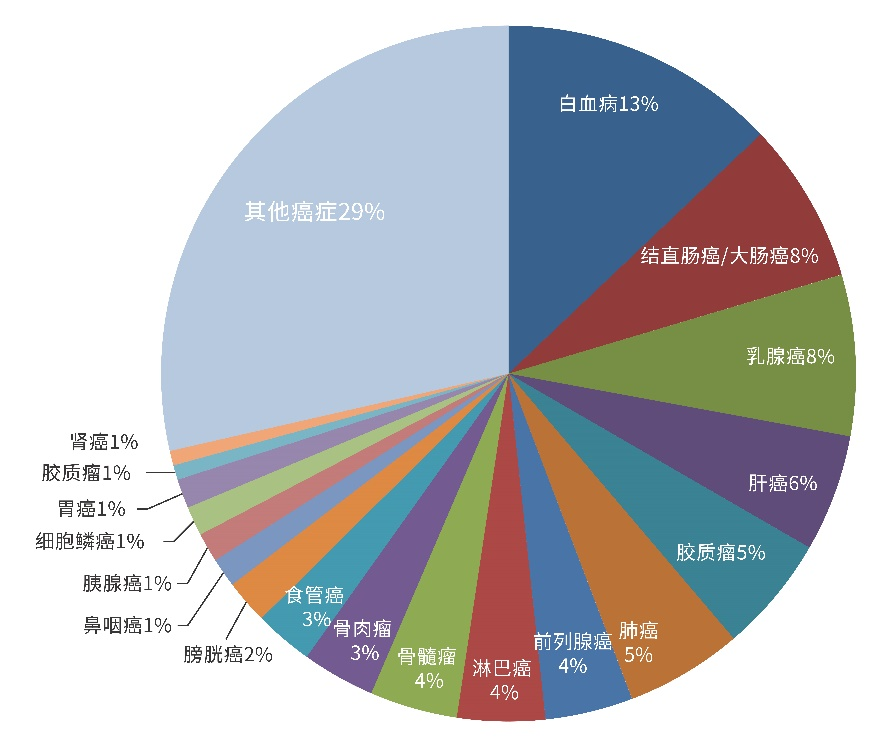
图3. 2020~2022年(截止6月)超级增强子肿瘤相关文章 (数据来源:Pubmed)
除了肿瘤以外,与超级增强子异常相关的疾病还包括有:阿尔兹海默症、类风湿关节炎、克罗恩病等。这些与免疫相关的疾病主要是由于T细胞产生了突变,而通过进一步的分析发现,这些突变位点大量分布于超级增强子区域内。
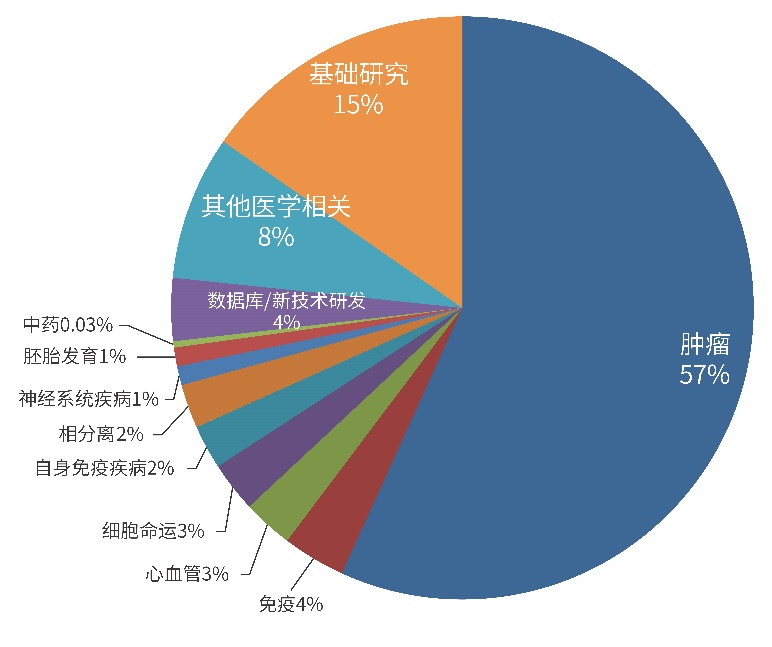 图4. 2020~2022年(截止6月)超级增强子相关文章研究方向 (数据来源:Pubmed)
图4. 2020~2022年(截止6月)超级增强子相关文章研究方向 (数据来源:Pubmed)
核心增强子hub enhancer
2018年,黄佳良研究团队分析Hi-C和ChIP-seq数据,发现超级增强子是由复杂的层次结构(hierarchy)组成的(点击查看文章解读)[3],由此定义了核心增强子(hub enhancer)和非核心增强子:先用标准的ROSE算法定义超级增强子;然后用层级评分(hierarchical score,简称H评分)给每个超级增强子的层级结构程度进行定量;随后,将每个超级增强子区间的染色质相互作用频率标准化,称为Z评分;第四,计算每个超级增强子所有区间中最大的Z评分,并参考这个超级增强子的H评分,高的H评分表明存在与超级增强子相关的染色质相互作用;第五,根据H评分的阈值,将所有超级增强子分成两类,一类是层级化的超级增强子,另一类是非层级化的超级增强子;最后,如果层级化的超级增强子(Hierarchical SEs)里的一个增强子元件,与一个Z评分高于H评分阈值的区间重叠,那么它就是一个核心增强子,这个超级增强子中的其他增强子就是非核心增强子。
层级化超级增强子的ROSE算法排名会比非层级化超级增强子更高,细胞类型特异的生物学过程相关GO注释会更丰富,比如K562细胞的会有血液凝固相关的注释,GM12878细胞会有B细胞稳态相关的注释。这些结果表明层级化的超级增强子在维持细胞身份方面具有重要作用。超级增强子的显著特征之一就是富含有细胞类型特异主要调控因子和激活因子,而核心增强子含有显著更高的层级结构调控主要因和激活因子,比如ChIP-seq结合信号、cohesin和CTCF等,表明核心和非核心增强子在活性增强子相关的组蛋白修饰和层级结构特异的转录因子方面有着一些不同点。
此外,核心增强子也富集了更多与疾病相关的遗传突变以及细胞类型特异的基因表达的遗传突变,提示核心增强子能够调控疾病发展,与疾病风险相关。
hub RNA又是什么?
2020年,薛愿超研究团队[4]利用RIC-seq技术绘制了HeLa细胞的转录组RNA 3D交互图,验证了2307个RNA拓扑结构域(点击查看文章解读)。通过对目标基因数量和相互作用强度进行划分,确定了642个RNA相互作用中心,命名为hub RNA。RNA 70%来源于含有大内含子(>50 kb)的编码基因, 只有5%来源于lncRNA和伪基因,而hub lncRNA表现出比 hub pre-mRNA更强的反式作用。Hub RNA与非hub RNA相比具有更强的反式作用并且其结构更为保守。
知识卡片:hub RNA的特征
(1)它们通常与来自同一染色体和亚细胞结构的目标RNA相互作用;
(2) 根据187种已知的RBP结合模体,hub RNA及其作用靶标可分为三类,来自相同类的hub RNA通常相互作用;
(3)在相同的GO term 中的hub RNA 不表现优先的相互作用;
(4)13%的hub RNA与超级增强子(SE)重叠,这些hub RNA作为SE富集到CCUUCCC模体,并被RBP所占据。
非编码区域突变与疾病
超级增强子存在于基因组中的非编码区域。现如今,科学家们越来越清楚地认识到非编码区域的突变对疾病的影响作用。
人类基因组测序计划研究表明蛋白质编码序列仅占基因组的2%,而剩余98%为非编码核酸序列,它们经过广泛地转录,产生了海量的非编码RNA分子,在复制、转录、翻译等遗传信息传递过程中发挥了关键的调控功能[5-6]。已知与癌症、心脑血管系统疾病、神经系统疾病、代谢性疾病和免疫系统疾病相关的突变约90%发生在非编码区,但是致病机理不明。
非编码RNA研究的难点在于,与蛋白质编码基因不同,非编码RNA不具有蛋白质编码潜能,其功能的发挥往往需要形成复杂的高级结构,进而在RNA结合蛋白(RNA-binding protein,RBP)的介导下通过与其他mRNA和非编码RNA分子相互作用而实现。因此,解析RNA分子的结构和相互作用位点成为理解其功能机制的关键,这也是RNA领域新技术发展的方向和难点。
利用高通量结构解析技术系统分析细胞分化、信号转导、个体发育过程中RNA结构的动态变化与基因表达和细胞表型的关系将是未来RNA领域研究的重点。此外,与重大疾病相关的非编码致病突变如何影响、改变RNA高级结构也是亟待解决的基本科学问题[7]。
随着对RNA结构功能性理解的深入,以RNA高级结构作为新靶点设计小分子药物有望成为疾病治疗的重要手段。此外,以RNA结构为基础,设计针对单链区的siRNA、ASO和LNA,必将加快核酸药物研发的步伐。
超级增强子的临床应用
超级增强子的先驱者Richard A. Young与JQ1/iBET研发者Bradner J.E.这两位科学家曾预言超级增强子具有广阔的研发前景和价值,必将成为下一个药物研发的黄金靶点,有望开发一种精确影响基因调控元件的药物。这两位科学家也因此联手成立Syros公司,研发针对超级增强子的抗癌药。经过了6年的发展,如今以超级增强子为靶点的肿瘤治疗用小分子抑制剂已进入临床研究阶段。
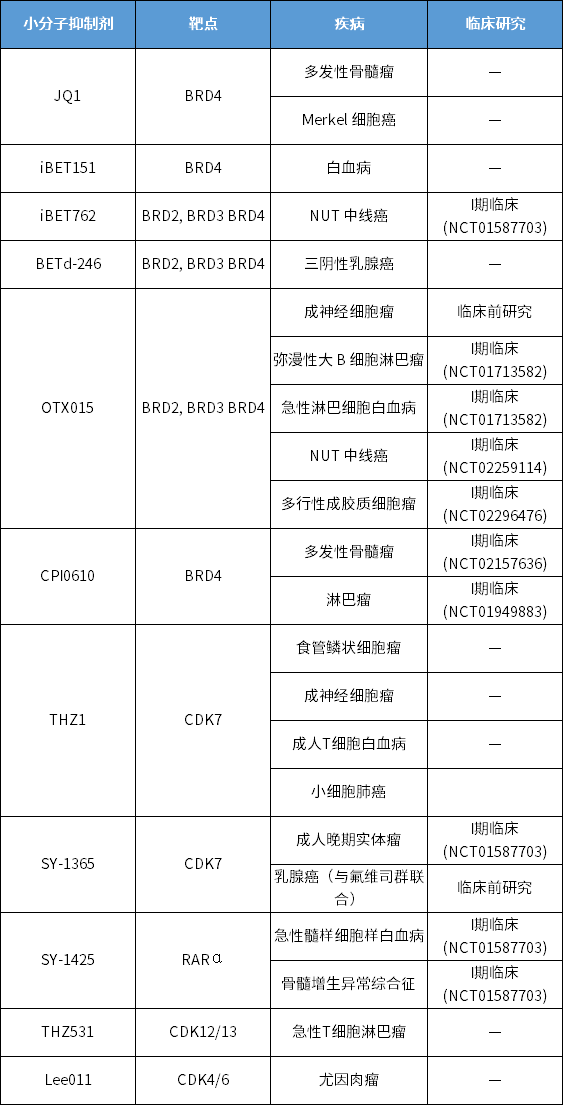
表 1 在肿瘤治疗中以超级增强子为靶点的小分子抑制剂研究现状[1]
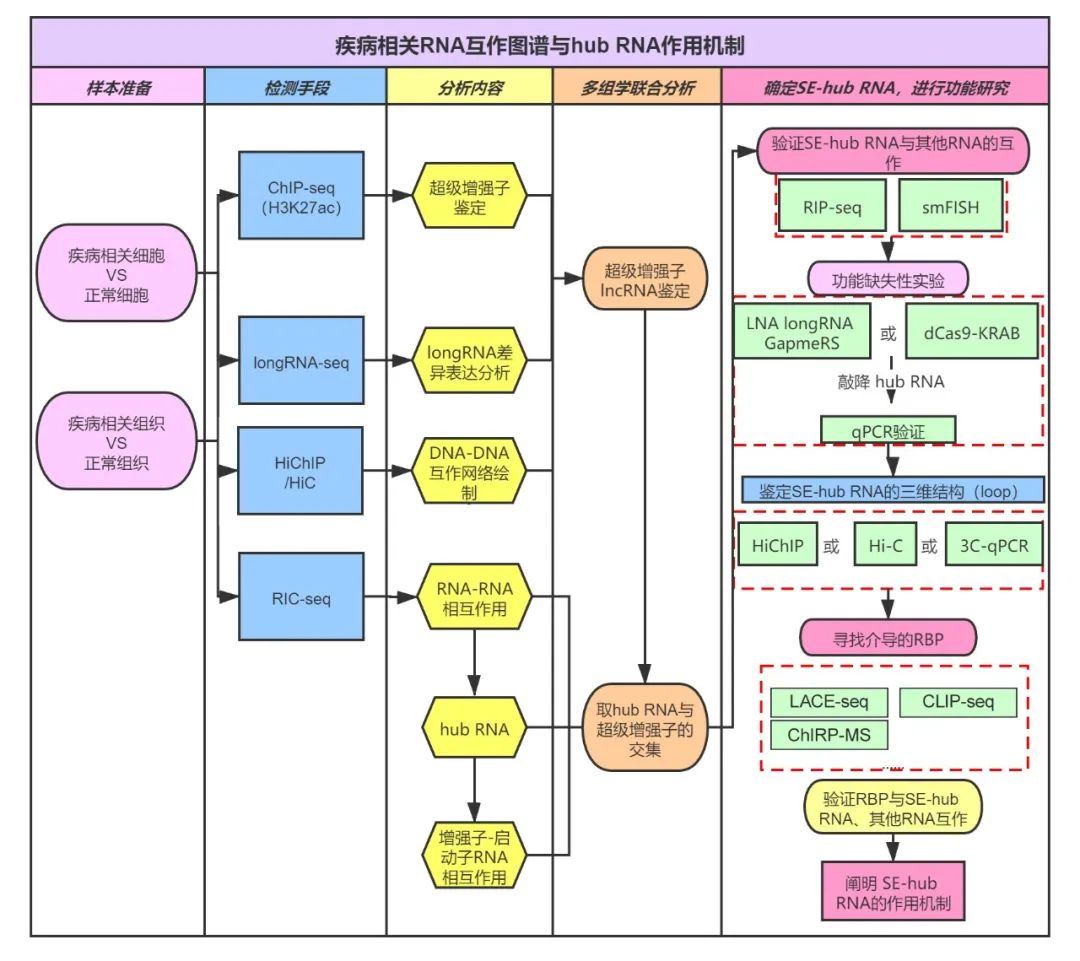
超级增强子研究从何入手?
关键技术:ChIP-seq,CUT&Tag,ATAC-seq
上面我给大家介绍了超级增强子相关的背景知识,接下来,该说说如何开展超级增强子研究了。首先,我们需要对超级增强子进行鉴定。
ChIP-seq即染色质免疫共沉淀测序,可确定蛋白质与DNA之间的相互作用。因此,利用针对增强子相关转录因子及其辅因子(如Mediator、BRD4)或组蛋白修饰(如H3K27ac、H3K4me1)进行ChIP实验,结合高通量测序,然后使用ROSE算法,可从ChIP-seq数据中鉴定超级增强子,这也是目前使用最多的超级增强子鉴定技术。
而在2019年,一个新的蛋白质-DNA互作关系研究方法出现了:CUT&Tag,即靶向剪切及转座酶技术,利用酶锚定技术绘制蛋白质—DNA互作图谱,和ChIP-seq一样使用抗体特异性结合目的蛋白,但不需要甲醛交联和免疫共沉淀,因而所需细胞量少,信噪比高。
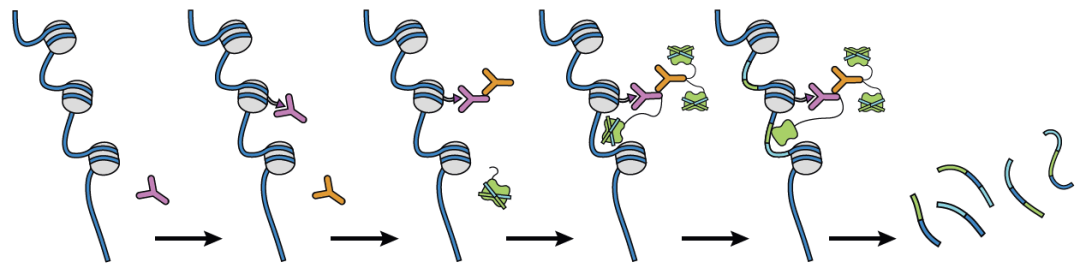
图5. CUT&Tag技术原理:将特异的抗体(如anti-H3K27ac)与染色质蛋白原位结合,然后锚定到A-Tn5转座酶融合蛋白。Tn5转座酶活性被激活,仅将抗体靶蛋白结合的染色质片段化,高效产生高分辨率、低背景的片段文库。
知识卡片:两种超级增强子鉴定技术,如何选择?
(1)根据细胞样本的细胞数量:在单个样本能达到1x107个细胞的时候,可以选择ChIP-seq,甲醛交联后的细胞沉淀,在干冰运输的条件下样本状态是相对稳定的;而细胞数量较少(20万个)的样本,可以选择CUT&Tag。
(2)根据样本类型:组织样本,可以选择CUT&Tag技术,但此技术对样本活性要求高,需要样本活性较好。 大家可依据自己样本的类型、细胞数量、活性状态选择合适的鉴定技术。
除此以外,假如不确定手上的课题是否涉及表观修饰,表观遗传学是否参与调控机制的时候,可选择高效探索染色质开放性的技术——ATAC-seq。ATAC-seq适用于探索表型变化、疾病等与基因调控的相关性,以其准确率高、重复性好、所需样本量少的特点,成为目前检测染色质开放性的最优策略。
将ChIP-seq或CUT&Tag数据进一步进行CRC(核心转录因子调控回路)分析,再加以longRNA-seq联合分析差异表达基因,可找到超级增强子潜在的靶基因,推动下一步的研究。
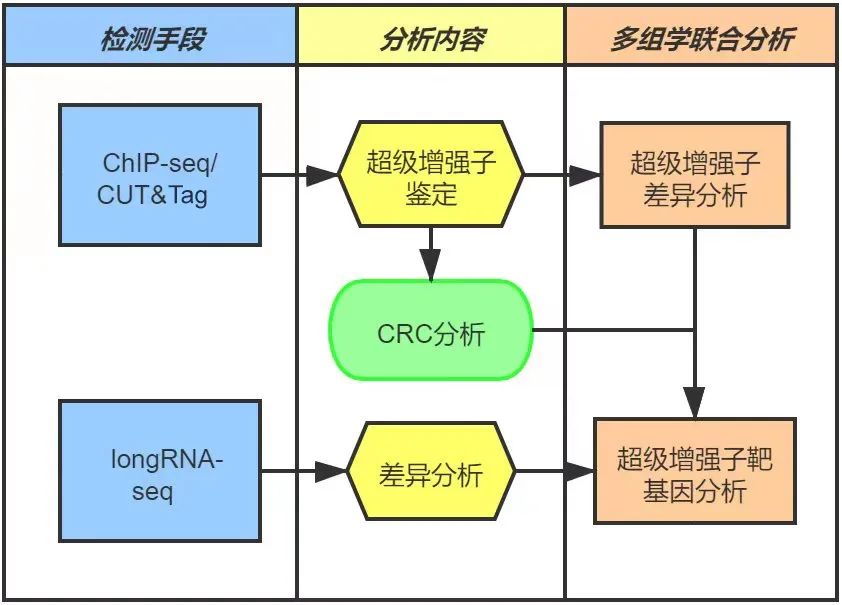
图6. 超级增强子鉴定部分研究路径归纳
增强子与启动子的互作很重要
关键技术:HiChIP,RIC-seq
我们已经知道,启动子决定转录的起始位点,而增强子决定一个基因在什么组织、什么时间发生转录。但是,增强子如何促进启动子的转录,是转录领域里面悬而未决的问题。
启动子和增强子在转录的过程中,要形成loop,使远距离的增强子和启动子,变得相近。不过,基因组里大概有大概有5万个启动子,而增强子至少有10万个,它们之间的对应调控关系是现代生物学研究的热点和难点。非编码基因发挥作用,往往通过基因间的相互作用,要深入研究非编码区,绕不开对相互作用靶基因的探索,所以在三级(高级)结构层面的全局基因互作图谱的绘制尤为重要。
探索DNA-DNA互作,我们可以利用HiChIP技术。HiChIP结合了Hi-C和ChIA-PET两个技术:ChIA-PET主要纯化手段只有染色质免疫沉淀,in situ Hi-C则只有生物素富集,而HiChIP则是将两者巧妙结合,所以比ChIA-PET有效信息读取率更高,所需细胞量更少。简单概括的说,HiChIP就是在原位产生Hi-C交联,利用生物素标记DNA末端,后续用特异性的抗体进行ChIP实验,最后再用生物素捕获Hi-C交联,进行测序。
以研究启动子和增强子的相互作用为例,我们可以用靶向目标转录因子的抗体(比如H3K27ac)将互作的启动子和增强子拉下来,进行测序,就可以获得增强子和启动子DNA互作图谱了,又通过启动子和靶基因的联系,推测增强子潜在靶基因,甚至预测hub enhancer。
检测DNA—DNA的互作有HiChIP;检测RNA—RNA的互作,则可以交给RIC-seq。
启动子和增强子区都可转录产生RNA,且增强子和启动子在空间上邻近配对后才能激活转录。这使得新开发的RIC-seq技术能够派上用场。RIC-seq (RNA in situ conformation sequencing)[4]技术可一次性捕获细胞内所有直接的RNA-RNA配对或者由蛋白质介导的间接RNA-RNA近距离相互作用,达到单碱基分辨率,不仅找到整个细胞内各种ncRNA的靶标,还能解析ncRNA的高级结构,这是目前RNA领域最大的难度,能实现Hi-C无法达到的功能。我们可以将RIC-seq用于系统鉴定非编码RNA的体内作用靶标,解密非编码RNA的高级结构、作用靶标和调控功能,预测hub RNA,为从RNA-RNA相互作用(比如启动子与增强子的互作:eRNA-uaRNA)角度理解非编码RNA突变的致病机理奠定基础。
知识卡片:RIC-seq技术应用
1. 在单碱基分辨率捕获miRNA前体,数量最多,最准确
2. 准确鉴定各类非编码RNA的作用靶标(lncRNA、snoRNA、rRNA等)
3. 准确捕获RNA的3D结构,包括cryo-EM技术忽略的RNA结构
4. RNA-RNA相互作用图谱
5. 鉴定Hub RNA
6. 发现增强子和启动子RNA相互作用
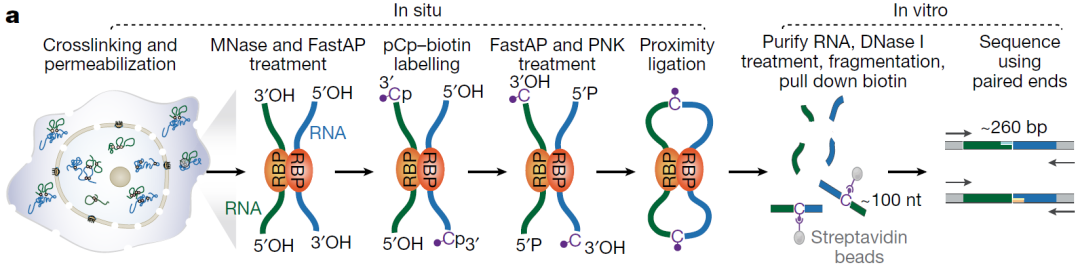
图7. RIC-seq技术流程:对细胞进行甲醛交联以固定蛋白质介导的RNA-RNA原位相互作用,之后在保持细胞完整性的情况下进行膜穿孔,并利用微球菌核酸酶处理以去除游离的RNA片段;然后在RNA的3'末端进行pCp-biotin标记并在原位进行近端连接;最后,裂解细胞,提取总RNA,纯化含有C-biotin标记的嵌合体RNA片段进行建库测序。
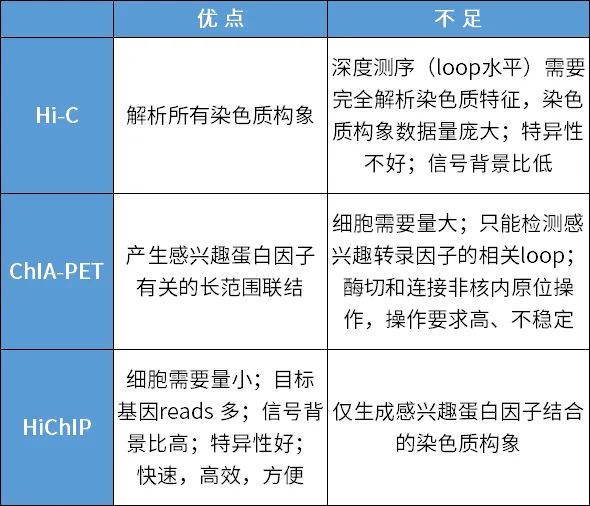
表2. Hi-C 、ChIA-PET 及HiChIP 技术的比较
在这里,我们还可以将第一步鉴定所得的超级增强子与HiChIP数据联合,分析超级增强子的靶基因,甚至hub enhancer;而RIC-seq定义的hub RNA本来就往往与超级增强子有重叠,将RIC-seq与超级增强子多组学联合分析,取交集的hub SE-hub RNA,可作为下一步开展分子实验,探究非编码区域致病突变基因调控机制的研究对象。
案例解读
薛愿超研究团队[4]通过RIC-seq鉴定得CCAT1是一个hub RNA,同时CCAT1与超级增强子638重叠,并且已被证明在多种癌症中上调;RIC-seq数据还显示它的异构体CCAT1-5L与MYC启动子RNA及PVT1 eRNA相互作用。于是研究者选取了super enhancer CCALT1-5L作为下游继续研究的基因。
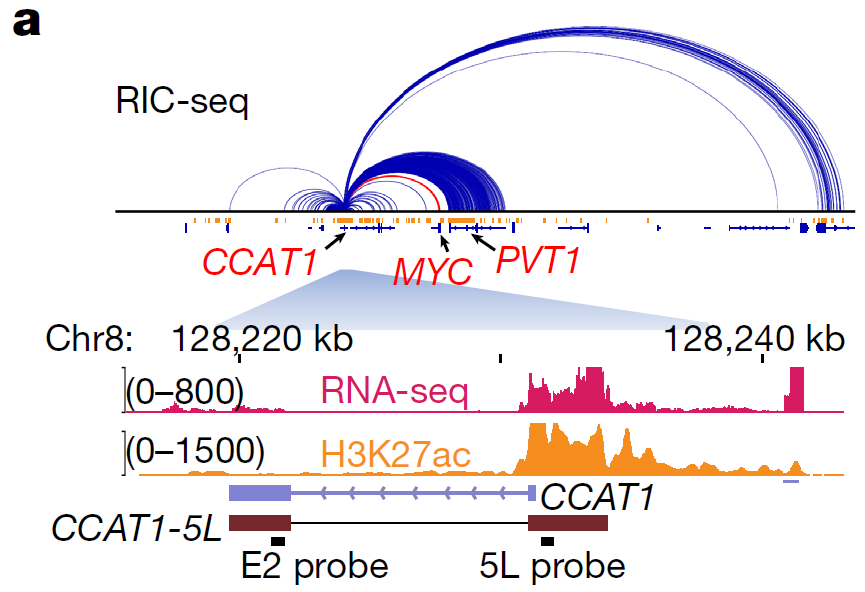
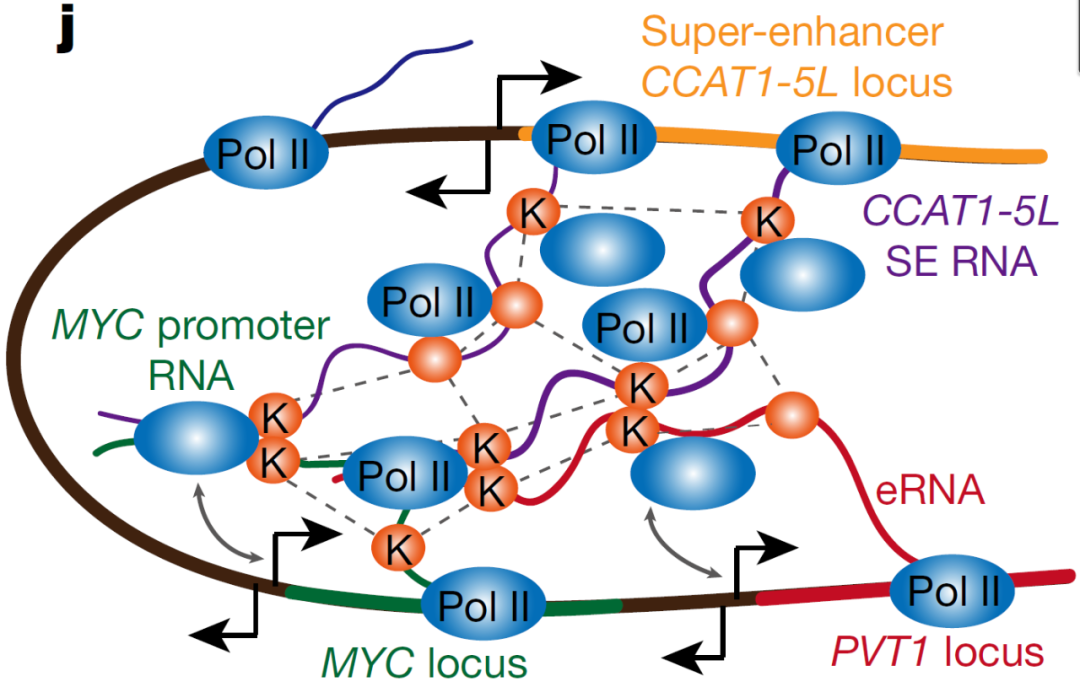
图8. CCAT1位点的RIC-seq、RNA-seq和H3K27ac信号,显示CCAT1-5L、MYC和PVT1相互作用
(在下文各个研究思路拆分部分,我都将以这篇薛研究员2020年发表于Nature的超级增强子hub RNA研究作为案例,让大家更好理解SE-hub RNA研究思路。)

图9. 增强子与启动子互作、敲定研究对象SE-hub RNA部分研究路径归纳
湿实验来啦:开展机制研究
1. 验证疾病相关SE-hub RNA的靶基因
关键技术:RIP-seq、smFISH
锁定了研究目标SE-hub RNA,接下来就是我们都不陌生的机制研究部分了。在这一步,我们可以利用smFISH(单分子荧光原位杂交)技术,进一步验证HiChIP预测的hub enhancer相互作用靶基因,或RIC-seq提示的潜在hub RNA与其他基因的相互作用,找到靶基因。RIP(RNA Immunprecipitation)是研究细胞内RNA与蛋白结合情况的技术,适用于研究转录后调控网络动态过程,我们可以利用RIP-seq技术研究与目的蛋白互作的RNA。
案例解读
薛研究团队使用smFISH实验显示hub RNA CCAT1-5L与MYC启动子RNA及PVT1 eRNA三者的共定位,这个证据支持了RIC-seq预测的CCAT1-5L与MYC启动子RNA和eRNA相互作用以调节远程染色质环的模型。
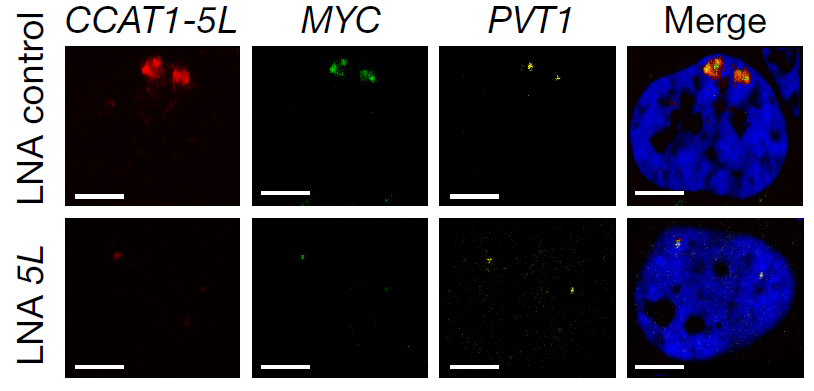
图10. smFISH显示hub RNA CCAT1-5L与MYC启动子RNA及PVT1 eRNA三者的共定位
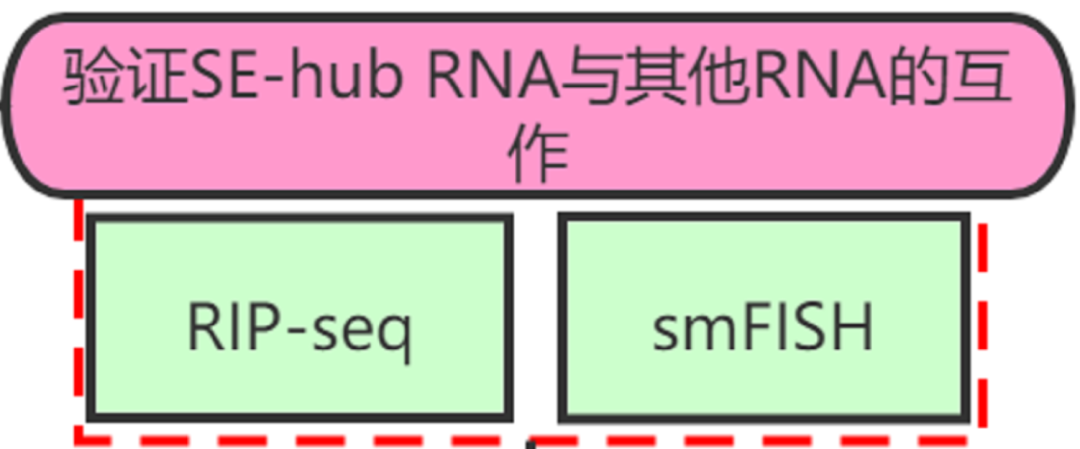
图11. 验证SE-hub RNA的靶基因部分研究路径归纳
2. 功能缺失性实验
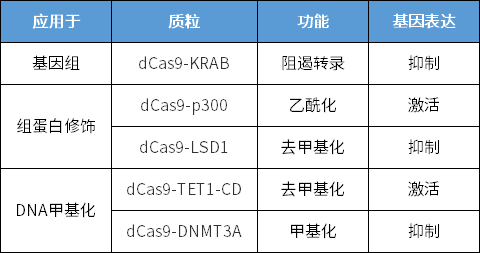
关键技术:LNA敲降,dCas9-KRAB/ dCas9-LSD1
在确定了可能与非编码区域致病突变相关的超级增强子或其靶基因之后,我们可以通过多种分子实验来验证其功能机制。最经典且可靠的,就是功能缺失性/获得性实验。
采用LNA longRNA GapmerRs敲降hub RNA的表达,或通过dCas9-KRAB/ dCas9-LSD1等抑制超级增强子,通过双荧光素酶报告基因实验检测增强子活性,qPCR技术验证下游调控基因的表达水平。
此外,还可以使用dCas9-p300的表观遗传激活系统,进行功能获得性实验。
表3. 基于CRISPR/dCas9的表观遗传激活和抑制系统
案例解读
在功能缺失性实验这部分,薛研究团队利用LNA敲降、CRISPR-Cas9敲除和CRISPR干扰等多种手段抑制CCAT1-5L后,MYC基因的转录显著下降。
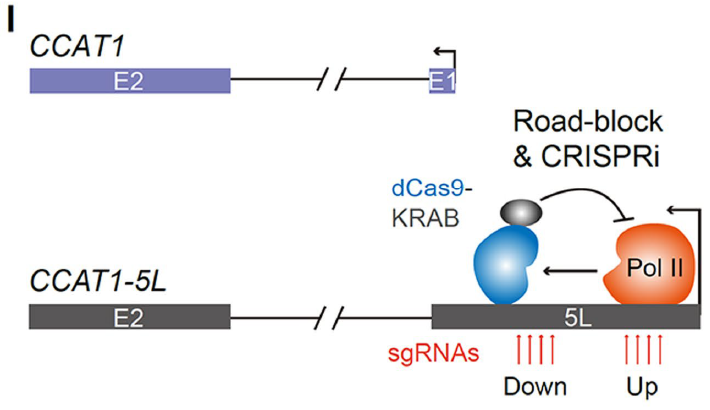
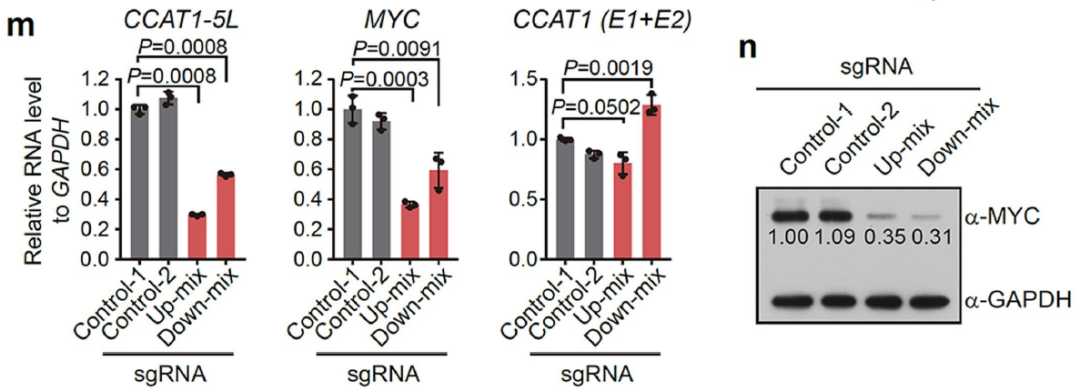
图12. l. dCas9-KRAB设计示意图;m~n. qPCR和Western blot结果显示抑制CCAT-5L之后,MYC表达下降
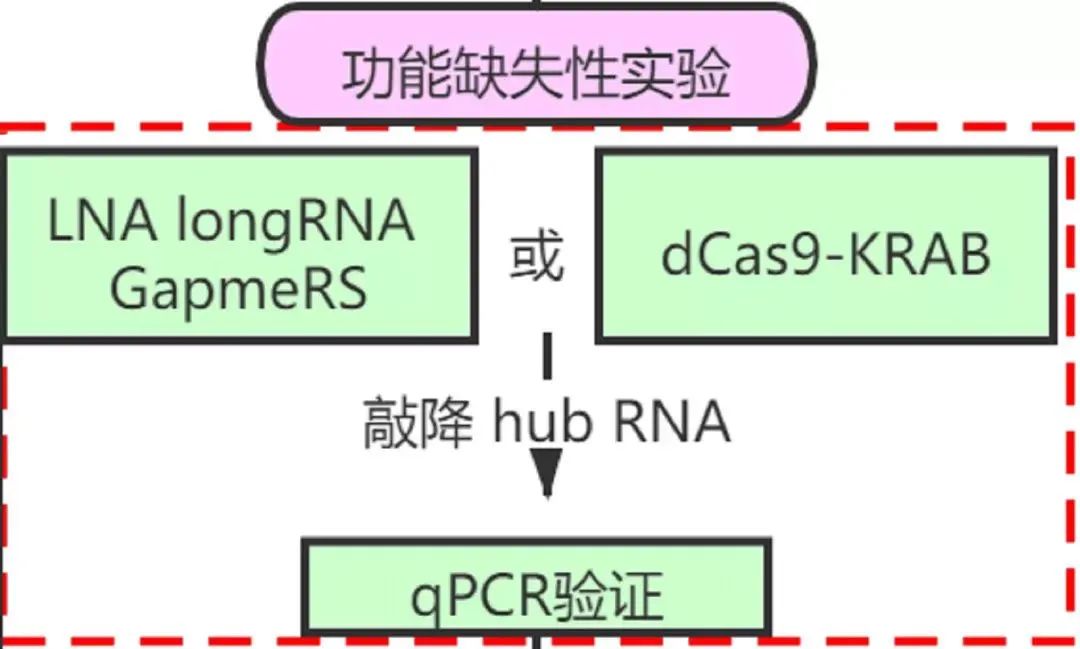
图13. 功能缺失性实验部分研究路径归纳
3. 鉴定SE-hub RNA的三维结构(loop)
关键技术:RADICL-seq,HiChIP-seq,Hi-C,3C-qPCR
前面RIC-seq所预测的三维结构,可以通过其他技术获得更多的证据,进一步验证。比如可以利用RADIBL-seq、HiChIP、Hi-C或3C-qPCR技术,验证hub RNA与启动子RNA、eRNA在三维结构上是否存在相互接触的空间条件(比如染色指环),实现远端(long-range)调节。
知识卡片:RADICL-seq
RADICL-seq是一种在全基因组范围内检测RNA与DNA相互作用的新方法,绘制细胞核内RNA与染色质的相互作用图谱,可应用于解析RNA分子介导的转录调控机制、构建三维基因组调控网络以及破解非编码区、增强子区突变所介导的调控失常
案例解读
薛研究团队使用3C-qPCR进行定量染色体构象捕获实验,显示CCAT1-5L、PVT1和MYC位点之间的染色质环在CCAT1-5L缺失时显著减少。这个数据支持了CCAT1-5L与MYC启动子RNA和eRNA相互作用以调节远程染色质环的模型,说明超级增强子RNA与启动子RNA之间的互作可调控染色质构象。

图14. 利用LNA敲降下调CCAT1-5L的表达后,MYC基因的转录显著下降,染色质构象捕获分析发现从CCAT1-5L超级增强子区到MYC启动子和增强子区的染色质环化效率也显著降低,说明超级增强子RNA与启动子RNA之间的互作可调控染色质构象
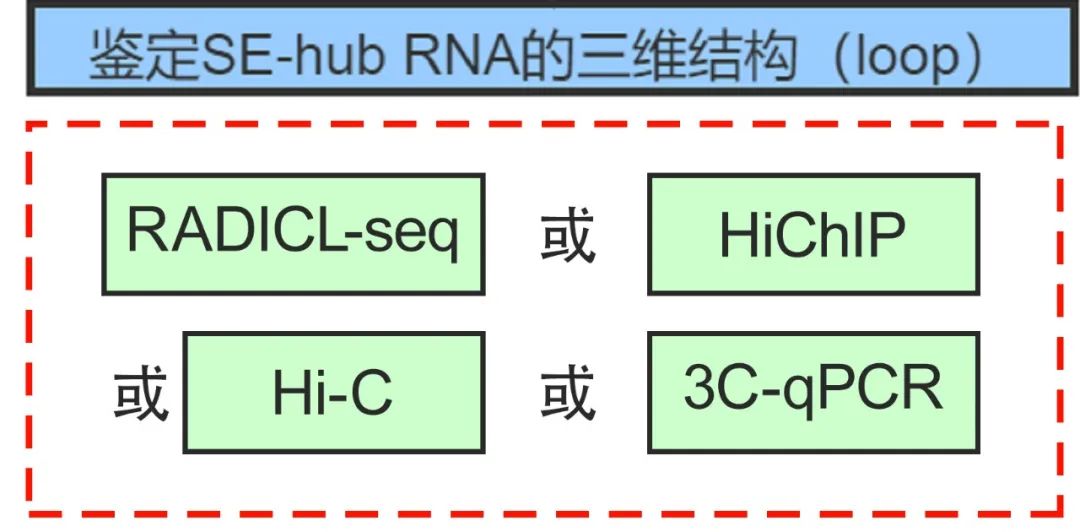
图15. SE-hub RNA三维结构鉴定部分研究路径归纳
4. 寻找介导调控的RBP,并从细胞水平验证
关键技术:LACE-seq,RIP-seq,CLIP-seq
人类基因组编码了约1500个RNA结合蛋白(RNA-binding protein, RBP),它们往往通过结合RNA分子上的特定基序或结构元件而调控细胞内各种RNA分子的加工、定位、翻译和稳定性等。在这一步,我们可以探索SE-hub RNA参与的致病机制中,介导调控的RBP,也就是这个hub RNA上的RBP,更深入的挖掘疾病相关SE-hub RNA所依赖的调控靶基因的机制。
目前常用的鉴定RBP靶标的方法主要有RIP-seq和CLIP-seq。不过这两种方法都依赖于特异性抗体富集RBP及其所结合的RNA,样本所需细胞数量也多。我们还有一个新的选择:LACE-seq。这是一项可在微量细胞中鉴定RBP作用靶点的新技术,它通过线性扩增逆转录酶在RBP结合位点处的终止信号鉴定RBP靶标,达到单碱基分辨率的精准鉴定。
通过LACE-seq技术,寻找hub RNA上的RBP;CLIP-seq进一步验证RBP与三种RNA是否存在相互作用。找到介导的RBP之后,至此,超级增强子hub RNA的基因调控机制已研究得十分深入了。
案例解读
薛研究团队为了解析SE-hub RNA CCAT1-5L激活MYC转录的详细分子机制,利用了ChIRP-MS、CLIP-seq、ChIP-qPCR等手段,发现RNA结合蛋白hnRNPK可与CCAT1-5L、MYC启动子RNA和增强子RNA PVT1相互作用,并通过coIP实验证实hnRNPK形成同型二聚体,并与RNA聚合酶II相互作用,把后者超级增强子区递送到MYC的启动子,从而促进MYC基因的转录。
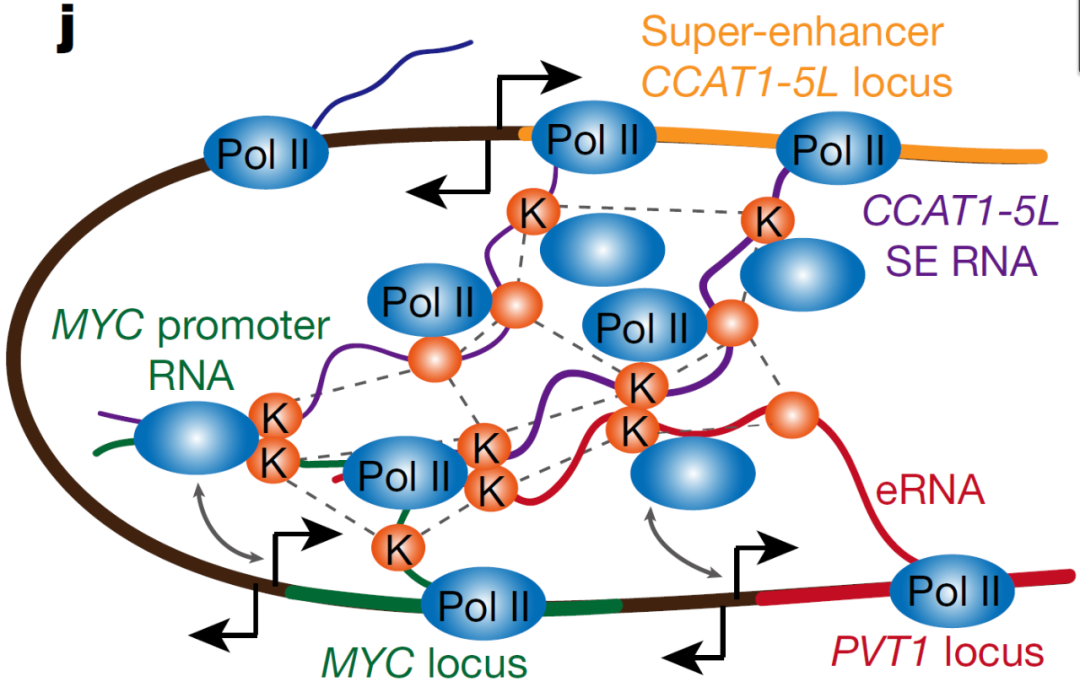
图16. 超级增强子通过染色质环(chromatin looping),激活MYC转录,从而调控MYC

图17. 寻找介导的RBP并验证部分研究路径归纳
写在最后
对于非编码基因的驱动作用,我们了解的太少,未知的还有太多。目前,我们的分析正在从外显子组转变为全基因组分析,意味着我们挖掘非编码区域调控驱动事件的能力正在不断提高。在这个领域找到新发现,为各种疾病发生发展提供更多信息,找到某种疾病发展相关的新的致病突变基因以及确定新的标志物,是一项重要的任务。然而,核心的挑战是从比编码基因组大50倍以上非编码基因组产生的大量非功能性“乘客突变”中分离出重要的“驱动突变”。可幸的是,我们拥有了更多不同检测技术,我们有着一以贯之的坚持,为在细胞和组织水平上寻找人类疾病发生发展中涉及的非编码区域驱动突变事件而努力。在这里,超级增强子hub RNA作为一种核心调控元件,将表观基因组和表观转录组跨界联合,有望成为一个助力非编码区致病突变研究的新兴方向,让我们拭目以待。
广州表观生物科技有限公司在国内率先推出完备的超级增强子研究解决方案,为研究者提供RIP-seq、HiChIP、超级增强子鉴定、RADICL-seq、LACE-seq等多项技术服务,目前已经帮助多个客户在这个领域取得丰硕成果。如果你对这个方向也感兴趣的话,欢迎咨询我们的科研顾问!

参考文献
[1] 吴志强, 米泽云. 超级增强子在肿瘤研究中的进展[J]. 遗传, 2019, 41(1): 41-51.
[2] Sabari BR, Dall'Agnese A, Boija A, et al. Coactivator condensation at super-enhancers links phase separation and gene control. Science. 2018 Jul 27;361(6400):eaar3958.
[3] Huang J, Li K, Cai W, et al. Dissecting super-enhancer hierarchy based on chromatin interactions. Nat Commun. 2018;9(1):943.
[4] Cai Z, Cao C, Ji L, et al. RIC-seq for global in situ profiling of RNA-RNA spatial interactions. Nature 2020 06;582(7812)
[5] Chen J, Xue YC. Emerging roles of non-coding RNAs in epigenetic regulation. Sci China Life Sci, 2016, 59: 227-35
[6] Xue YC, Chen RS, Qu LH, et al. Noncoding RNA: from dark matter to bright star. Sci China Life Sci, 2020, 63:463-8
[7] 蔡兆奎,薛愿超. RNA结构解析技术研究进展[J]. 生命科学. 2021(03)