SELETC-m6A单位点修饰定量检测技术试剂盒和技术服务

项目简介
SELECT-m6A修饰定量检测技术
SELECT(single-base elongation-and ligation-based qPCR amplification method)[1]检测技术,即单碱基延长和连接的qPCR扩增技术,只需三小时就能完成低丰度转录本中单碱基分辨率的m6A检测,无需抗体富集就能够针对性地对特定位点进行 m6A修饰定量,样本间修饰丰度差异分析以及鉴定目标位点是否存在m6A修饰。表观生物将隆重推出多款SELECT检测试剂盒,填补市场上特定位点m6A 常规检测技术的空白,协助研究者鉴定m6A相关蛋白的作用靶点,深入挖掘m6A修饰的作用机制,有望为RNA m6A修饰分子标志物的发现带来新突破。
应用与优势
技术应用
1. 验证样本间特定位点m6A修饰丰度差异
2. 检测RNA目标位点是否存在m6A修饰
3. 对特定位点的m6A修饰丰度进行绝对定量
4. 鉴定m6A修饰相关蛋白的作用靶点
技术优势
1. 荧光定量PCR进行精确定量,达到单碱基分辨率
2. 无需抗体IP,无需同位素标记,降低成本,可操作性高
3. 灵敏度高,总RNA所需样本低至1μg
送样要求
≥1μg/样本
总RNA
≥2.5×105个细胞/样本
细胞
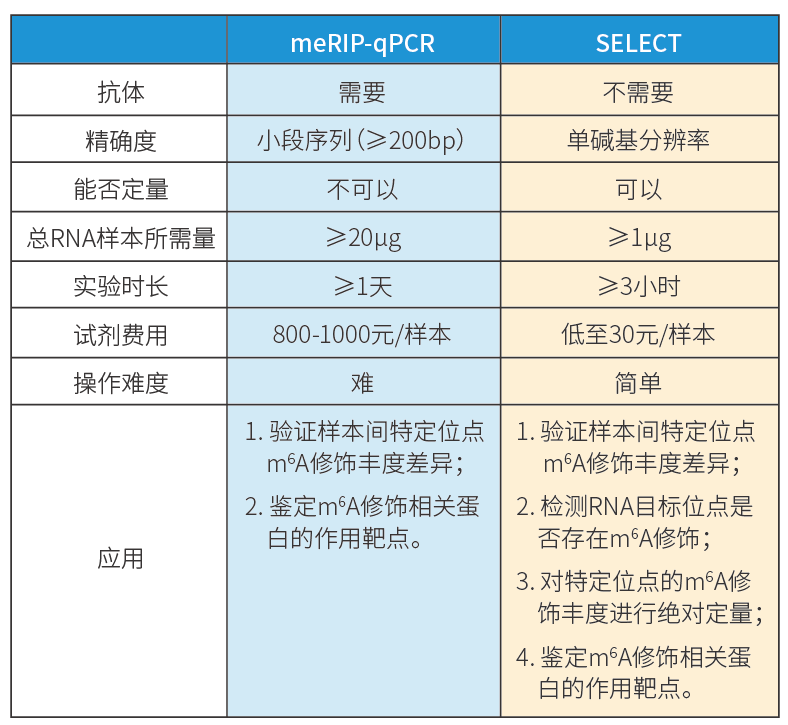
技术对比

试剂盒信息
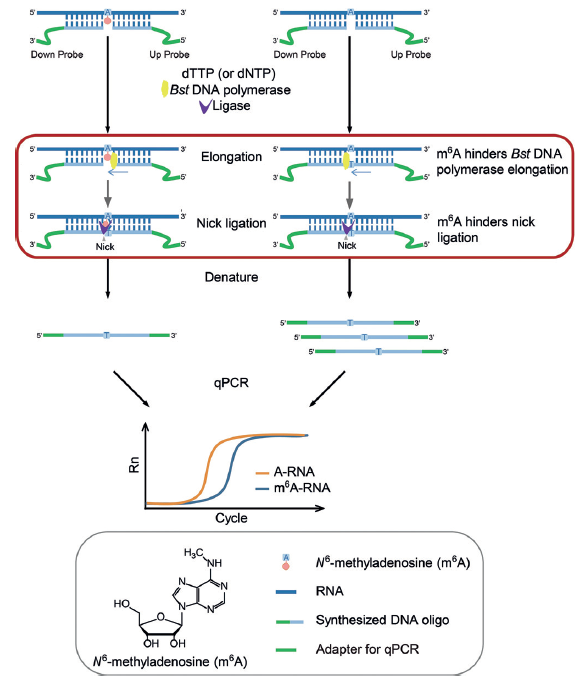
技术流程图
应用示例
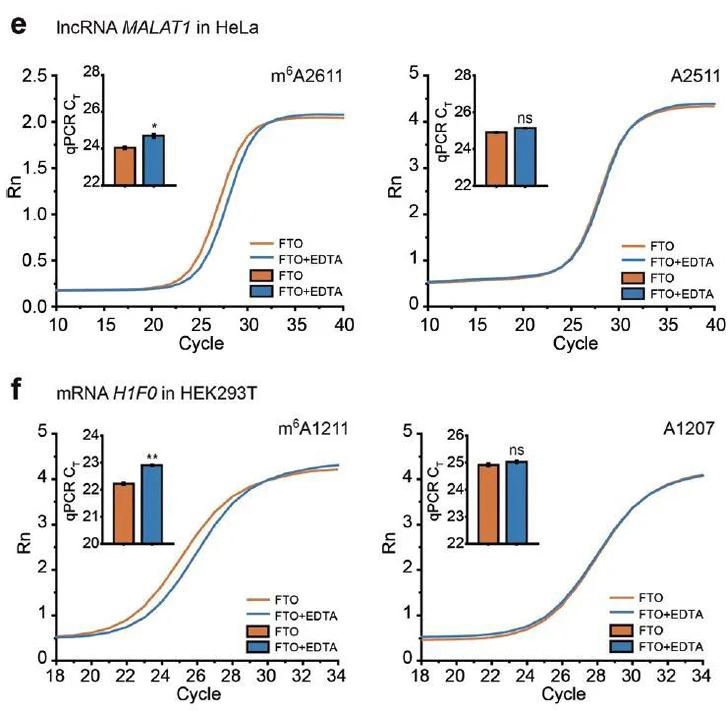
1. 检测RNA目标位点是否存在m6A修饰
图1. qPCR扩增曲线和CT值柱形图显示,
HeLa细胞lncRNA MALAT1 (e)以及HEK293T
细胞mRNA H1F0(f)的m6A位点和非m6A位点[1]
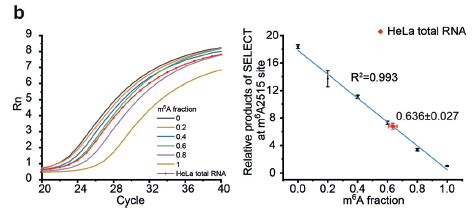
2. m6A对特定位点的m6A修饰丰度进行绝对定量
图2. 利用系列浓度梯度的已知m6A成分标准品制作标准曲线,然后对特定位点上的m6A修饰成分进行定量[1]
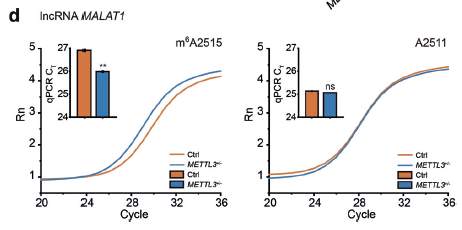
3. 鉴定m6A修饰相关蛋白(甲基化酶、去甲基化酶、 识别蛋白等)的靶点
图3. SELECT分析 lncRNA MALAT1和以及METTL3+/-细胞(对照组)的m6A2515和A2511(input对照)位点,荧光扩增曲线和qPCR CT值柱状图显示lncRNA MALAT1的2515位点是METTL3的一个靶点[1]
参考文献
[1] Xiao Y, Wang Y, Tang Q, et al. An Elongation- and Ligation-Based qPCR Amplification Method for the Radiolabeling-Free Detection of Locus-Specific N6 -Methyladenosine Modification. Angew Chem Int Ed Engl. 2018 Dec 3;57(49):15995-16000.
[2] Wang Y, Xiao Y, Dong S, et al. Antibody-free enzyme-assisted chemical approach for detection of N 6-methyladenosine[J]. Nature chemical biology, 2020, 16(8): 896-903.
[3] Liu X M, Zhou J, Mao Y, et al. Programmable RNA N 6-methyladenosine editing by CRISPR-Cas9 conjugates[J]. Nature chemical biology, 2019, 15(9): 865-871.
[4] Xu W, Li J, He C, et al. METTL3 regulates heterochromatin in mouse embryonic stem cells[J]. Nature, 2021, 591(7849): 317-321.
