SLAM-seq服务(RNA代谢测序)

项目简介
SLAM-seq服务(RNA代谢测序)
传统的转录组测序技术,可以揭示RNA在一个稳定状态下质和量上的变化,但是这仅仅是基因表达的一个“快照”,无法观察到RNA转录、加工和降解的细胞内动态过程。SLAM-seq,即thiol(sh)-linked alkylation for the metabolic sequencing of RNA,可以理解为“S4U烷基化RNA代谢测序技术”,可观察转录的实时情况和特征。
通过研发成熟的代谢RNA标记方法和高通量测序技术,SLAM-seq能从单核苷酸水平上检测整合到总RNA中的核苷类似物4-硫尿苷(S4U),绘制RNA聚合酶II依赖性的基因表达动态,是一种操作性强、稳定、可靠的基因表达调控机制研究方法。
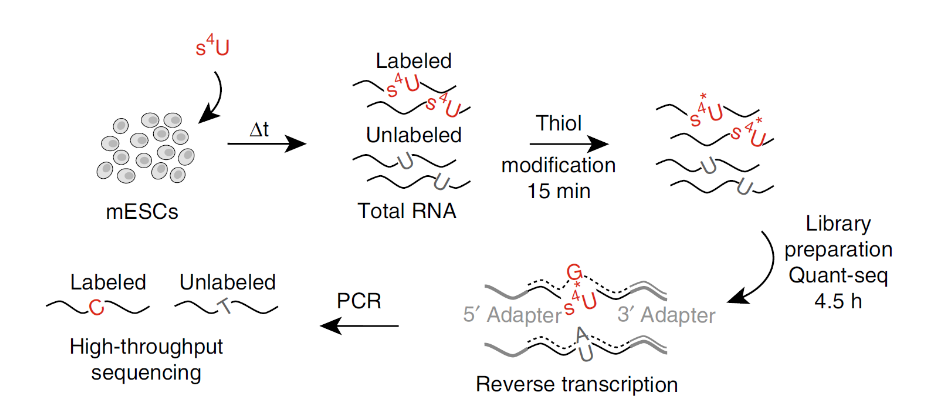
SLAM-seq技术流程图
技术原理
向合成中的RNA链中掺入4-thiouridine (S4U),使原本的碱基T被S4U修饰,从而在反转录中被错认为C,导致原本应该为碱基A的位置错配成为G。通过测序分析这些错配的G,就可以对新合成mRNA或其他longRNA进行直接的定量。
应用与分析
技术应用
1. 检测基因转录水平的变化
2. 研究RNA半衰期、稳定性
3. 深入研究基因转录调控机制
4. RNA修饰如m6A、m1A、ac4C等作用机制的研究
分析内容
1. 序列比对
2. 文库质控
3. Mapped Reads过滤
4. 背景T>C SNP校正
5. mRNA或longRNA*异表达分析
6. 靶基因GO富集分析
7. 靶基因KEGG富集分析
8. mRNA或longRNA转录本水平T>C counts计数与表达量化
9. mRNA或longRNA转录本水平Total reads计数与表达量化
10. mRNA或longRNA稳定性分析
11. mRNA或longRNA半衰期计算
送样要求
需要在培养的活细胞中掺入S4U碱基,可免费提供
1x10^6
细胞数量
人、大小鼠
样本物种
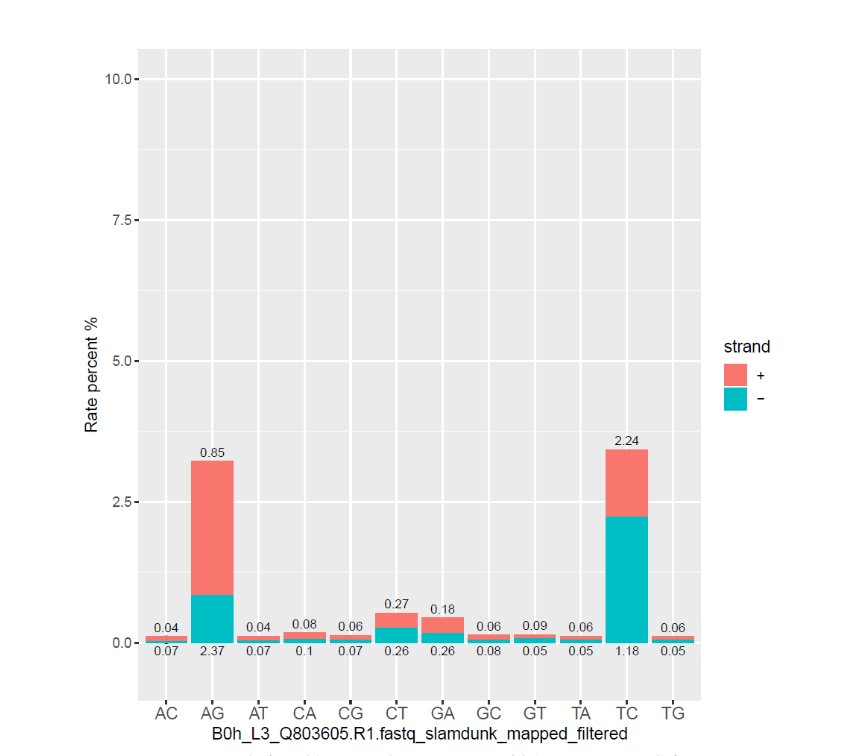
表观生物实测数据1
Slamseq建库质控(检测T>C和A>G转化是否明显升高)
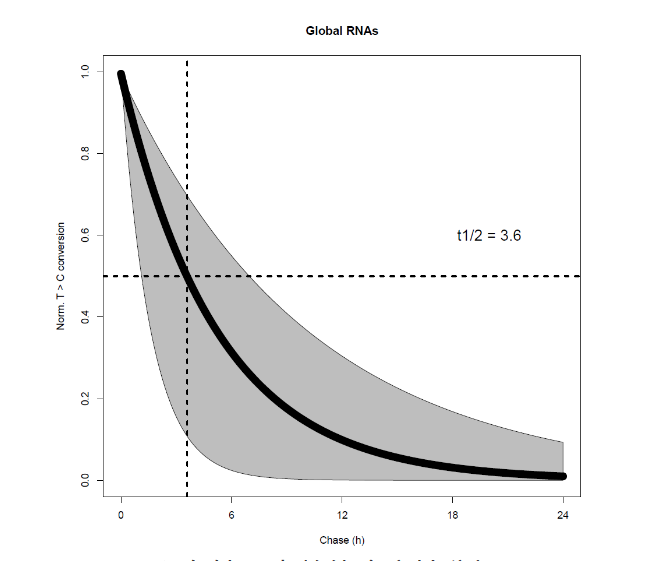
表观生物实测数据2
所有转录本整体稳定性分析

表观生物实测数据3
不同类型转录本稳定性比较分析
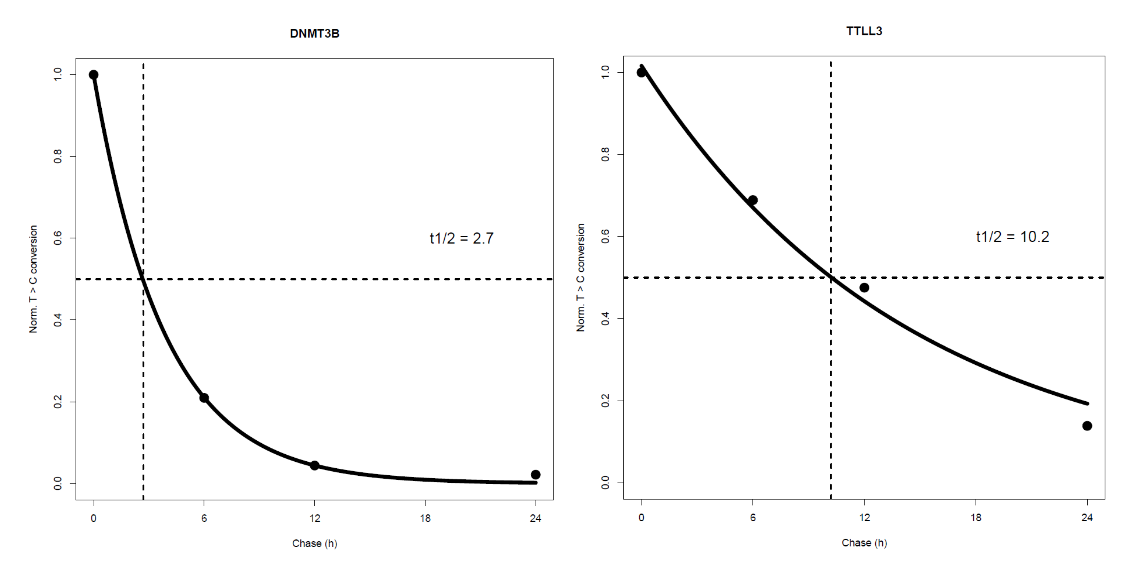
表观生物实测数据4
单个基因的稳定性分析(2个例子)
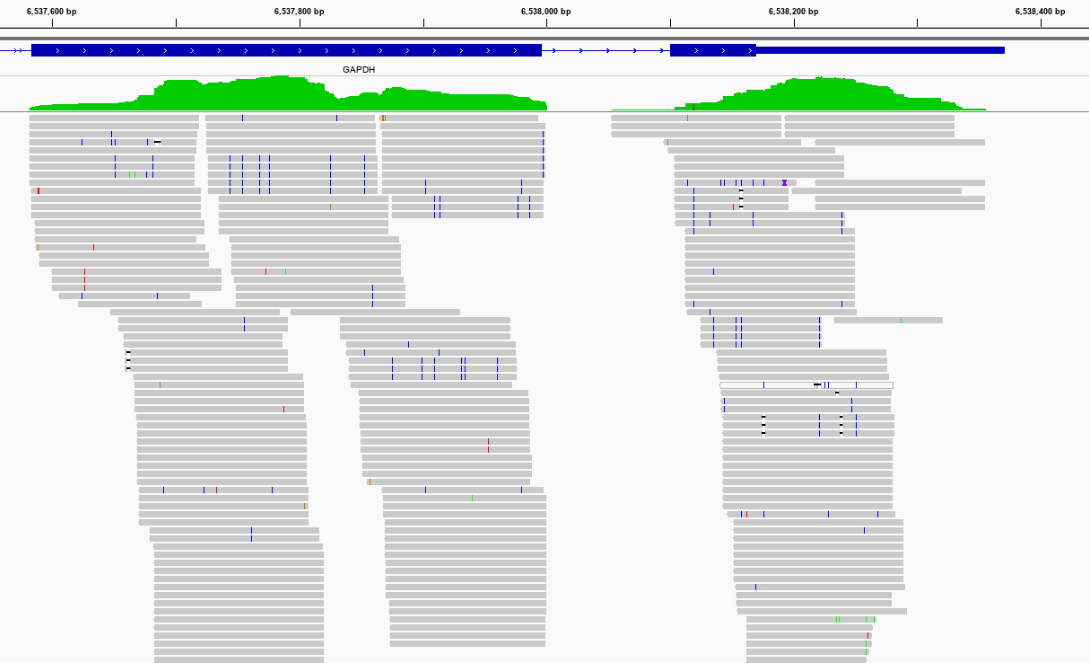
表观生物实测数据5
单个基因T>C突变查看(蓝色为T>C突变的碱基)
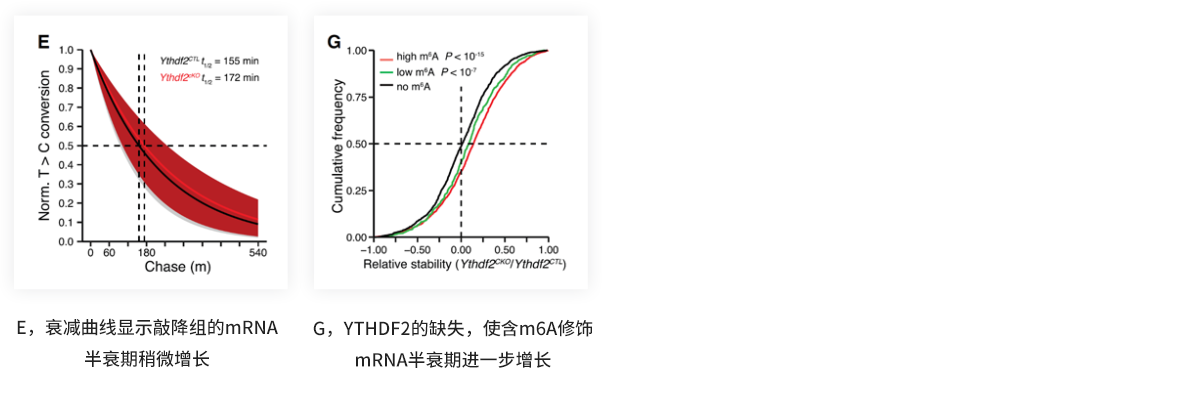
参考案例
本研究发现m6A阅读蛋白YTHDF2在AML患者身上广泛过表达,在小鼠和人类AML发生和发展过程中起着关键的作用。研究者敲除白血病前期细胞的YTHDF2,再进行meRIP-seq,发现敲除组中表达水平显著上调的mRNA具有丰富的m6A修饰。为了研究带有m6A修饰的mRNA的上调机制,研究者利用SLAM-seq检测mRNA的半衰期,证实m6A修饰使这些基因的半衰期延长了。
参考文献
[1].Herzog V A, Reichholf B, Neumann T, et al. Thiol-linked alkylation of RNA to assess expression dynamics[J]. Nature methods, 2017, 14(12): 1198.
[2].Paris J, Morgan M, Campos J, et al. Targeting the RNA m6A Reader YTHDF2 Selectively Compromises Cancer Stem Cells in Acute Myeloid Leukemia[J]. Cell stem cell, 2019.
