RADICL-seq
项目简介
RADICL-seq(RNA And DNA Interacting Complexes Ligated and sequenced)是一种探索 RNA 与染色质相互作用
的新技术,绘制细胞核内 RNA 与染色质的相互作用图谱,能够鉴定不同类转录本的基因组覆盖模式,以及细胞特异性的
RNA- 染色质相互作用。利用 RADICL-seq 可以在 RNA 与基因组靶标区域相互作用时捕获 RNA,并对捕获的每个 RNA 进行
全面定位。
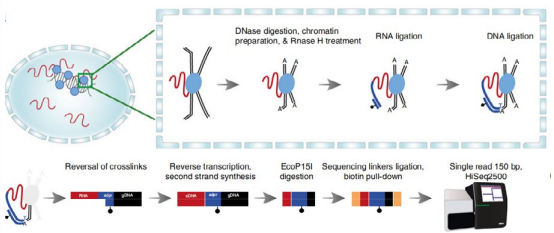
图 3-16. RADICL-seq 技术流程 [4]
技术原理
RADICL-seq 流程包括用甲醛固定细胞核内存在的 RNA、蛋白质和 DNA,然后通过独特设计的接头连接 RNA 和
DNA。之后去除固定的蛋白质,仅提取结合到衔接子的 RNA 和 DNA。通过下一代基因测序器读取这些基因,然后将其定
位到基因组中。
RADICL-seq 方法的主要步骤如下:借助 1% 甲醛处理细胞使之交联固定,然后分离细胞核,通过用 DNAseI 酶部分消
化基因组 DNA;再用 RNase H 处理消化核糖体 RNA 以提高结果的准确性;用接头将相邻的 DNA 和 RNA 连接起来,去交
联化后,将 RNA-DNA 嵌合体转化为双链 DNA,再进行测序。
应用与分析
技术应用
1. 系统鉴定细胞核内全基因组范围 RNA-DNA 相互作用
关系
2. 解析 RNA 分子介导的转录调控机制
3. 构建三维基因组调控网络
4. 破解非编码区增强子突变所介导的调控失常
送样要求
≥ 2×106 个细胞 / 样本
样本类型:甲醛交联细胞,
分析内容
1. 测序数据及其质量控制
2. Linker 比对
3. RNA/DNA 拆分及数据质控
4. 基因组比对
5. RNA-DNA 互作矩阵
6. 全基因组 RNA-DNA 互作绘图
7. RNA-DNA 虚拟 4C 绘图(高级分析)
8. RNA-DNA 互作增强子网络(高级分析)
图 1. 总体质控图
表观生物实测数据
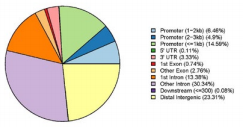
图 1. rna_pair_pie
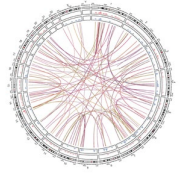
图 1. RNA-DNA 环状互作图
图 1. WT375_hic
参考案例
Bonetti A, et al. Nat Commun,2020. RADICL-seq 技术可在 RNA 与基因组靶标区域相互作用捕获 RNA[5]
RADICL-seq 技术基于临近位置的交联连接,与现有方法相比可以减少对新生转录本的偏向性、同时提高基因组覆盖率
和独特的绘图效率,可识别不同类别的转录本“占据”基因组的特有模式,以及识别细胞类型特异的 RNA- 染色质相互作用,
并揭示转录在染色质结构形成中的作用。该方法获得的数据可用于检查染色质如何组织和调节,并可全面评估 lncRNA 功能。

图 3-17. B. 对比 3 个重复样本的数据,RADICL-seq 显示出较高的重现性。E&F. 与 RNA-seq 对比,RADICL-seq 能捕获更多的核信息。E. 细
胞核的 RNA-seq 读数与 RADICL-seq 读数;F. 细胞质的 RNA-seq 读数与 RADICL-seq 读数。
1. 鉴定细胞核中的 RNA-DNA 相互作用,捕获更多的核信息
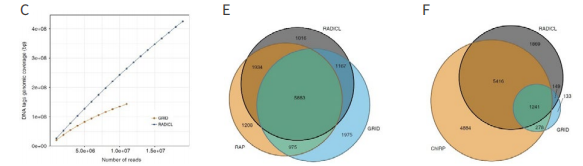
图 3-18. C. 与 GRID-seq 相比,RADICL-seq(蓝色)基因组覆盖率更高;E. 对比多种技术的 Malat1(一种 lncRNA)数据,RADICL-seq 能
检测到更多的靶 DNA;F. 对比多种技术的 Rn7sk(一种小核 RNA)数据,RADICL-seq 能检测到更多的靶 DNA。
2. RADICL-seq 分辨率更高,样本量更低,捕获率更高
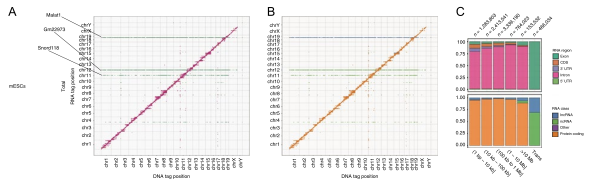
图 3-19. A&B. mESC 细胞 RNA-DNA 相互作用的二维相互作用矩阵图;C. 统计 RNA-DNA 相互作用发生的 RNA 区域和 RNA 类型
3. 鉴定全基因组的 RNA-DNA 相互作用,数据丰富可与 Hi-C 媲美
