RNA与RNA相互作用 RIC-seq
项目简介
RIC-seq(RNA In situ conformation sequencing)是一种能在细胞原位水平上捕获RNA高级结构及分子之间相互作用位点的新技术。RIC-seq能够对细胞中mRNA和非编码RNA的构象和组织规律,绘制全基因组增强子-启动子调控网络图谱,并阐明增强子激活癌基因转录的新机制。利用 RIC-seq 技术还可系统分析重大疾病相关突变对 RNA 高级结构和作用靶标的影响,这将有望揭示非编码区突变的致病机理,并为临床诊断和治疗奠定基础。

图1. RIC-seq技术流程[1]
技术原理
RIC-seq技术的基本流程是对细胞进行甲醛交联以固定蛋白质介导的RNA-RNA原位相互作用,之后在保持细胞完整性的情况下进行膜穿孔,并利用微球菌核酸酶处理以去除游离的RNA片段;然后在RNA的3'末端进行pCp-biotin标记并在原位进行近端连接;最后,裂解细胞,提取总RNA,纯化含有C-biotin标记的嵌合体RNA片段进行建库测序。
应用与分析
技术应用
1.鉴定细胞核内rna-rna相互作用
2.系统分析核内非编码RNA的高级结构和作用靶标
3.绘制RNA分子高级结构
4.构建RNA三维作用图谱
5.揭示非编码区突变的致病机理
基本分析
1. 序列比对分析
2. RNA表达分析及互作频率排序
3. 构建互作组矩阵
4. 增强子-启动子调控网络构建
5. 差异互作关系分析
高级分析
1. 差异互作与差异表达基因关联分析
2. 差异互作区域或差异互作RNA与GWAS关联分析
3. 三维基因组调控网络图绘制
4. 特定RNA 分子高级结构绘制
送样要求
≥1×10^7个细胞/样本
样本类型:活细胞
仅限人、大小鼠,其他物种需评估
样本物种
案例
Cai Z, et al. Nature, 2020. RIC-seq技术绘制细胞内RNA-RNA的原位三维作用图谱[1]
RIC-seq技术发现增强子和启动子非编码RNA之间的相互作用,可用于推断其调控网络,并详细解析超级增强子lncRNA CCAT1-5L与RNA结合蛋白hnRNPK、MYC启动子和增强子RNA结合以改变染色质构象,进而调控癌基因MYC转录的新机制。该技术不仅检测RNA的高级结构,而可鉴定各类非编码RNA的作用靶标,为后续深入研究非编码RNA及其功能性提供全新的新技术。此外,该技术可以应用于病毒RNA的结构与靶标的研究。
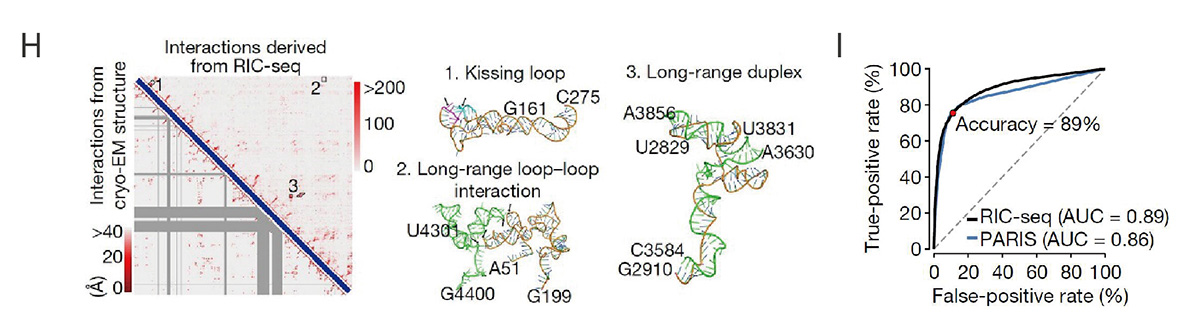
1. 捕获rRNA和lncRNA的3D结构
图2. H. 将基于RIC-seq的3D互作图谱与28S rRNA12的冷冻电镜(cryo-EM)模型进行对比。深灰色标记的区域为冷冻电镜中未检测到的互作区域。Box1、2和3分别对应右图不同类型的RNA-RNA相互作用。I图:ROC分析显示RIC-seq检测28S rRNA结构的性能,正确率为89%。
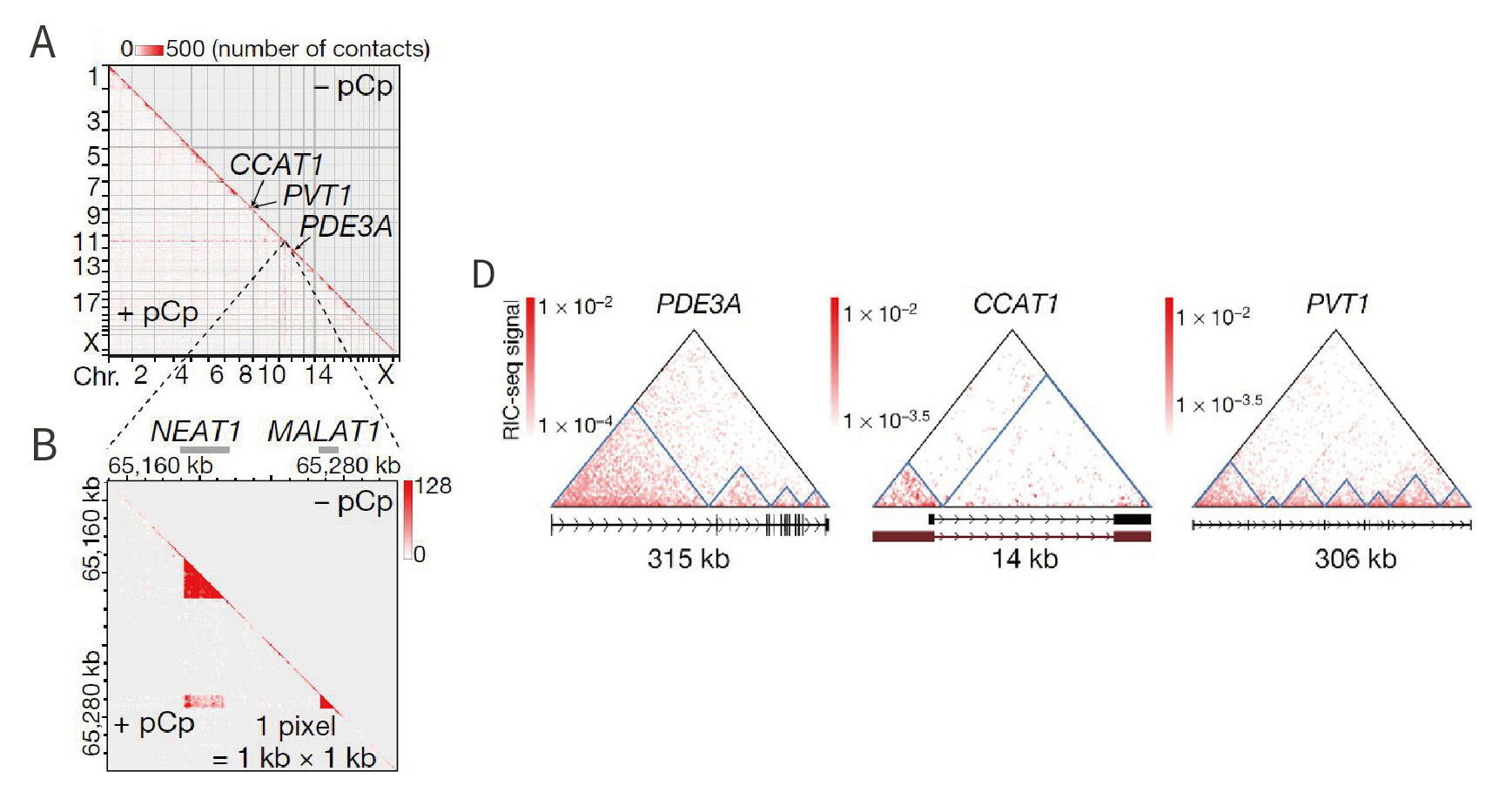
2.全局RNA互作图谱
图3. A. 从HeLa细胞的RNA 3D图谱中,作者鉴定出2307个RNA拓扑结构域,进而鉴定出642个“hub RNA”,包括lncRNA NEAT1 和MALAT1,表现出广泛的反式互作。
D. RIC-seq鉴定出3种RNA前体的拓扑结构域。
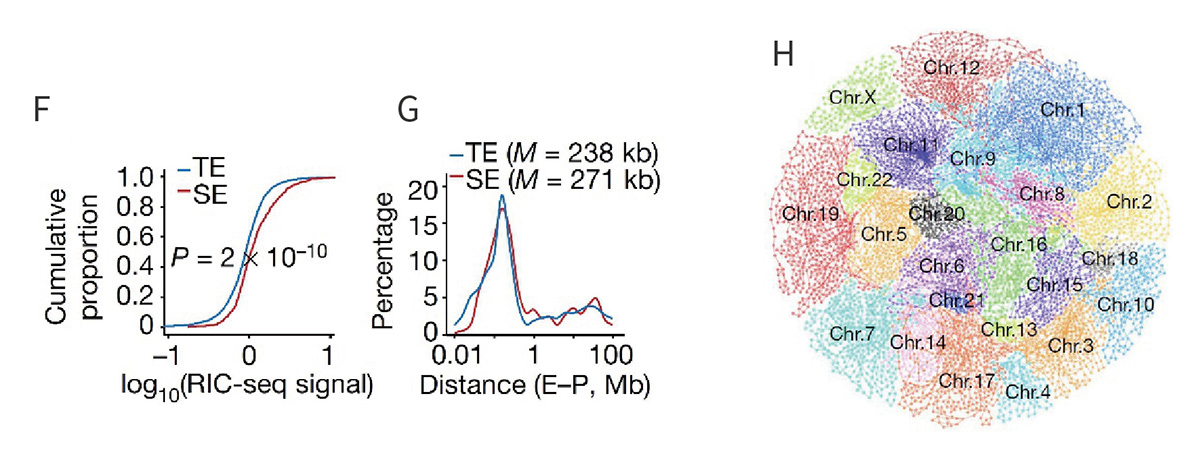
3. 增强子-启动子RNA相互作用图谱
图4. F&G. 超级增强子具有更强的RIC- seq信号,并且比普通增强子跨越更长的距离与其他RNA相互作用;H. 利用Cytoscape展现RIC-seq检测到的全基因组增强子-启动子和启动子-启动子网络。
参考文献
[1] Cai Z, Cao CC, Ji L. et al. RIC-seq for global in situ profiling of RNA-RNA spatial interactions. Nature,2020, 582, 432-437.
