TCGA数据挖掘服务| 肿瘤相关表观遗传调控基因深度分析
TCGA(The Cancer Genome Atlas),即癌症基因组图谱项目,始于2005年,由美国政府出资,美国国家癌症研究所(National Cancer Institute)和人类基因组研究所(NationalHuman Genome Research Institute)共同监督,旨在应用高通量的基因组分析技术,以帮助人们对癌症有个更好的认知,从而提高对于癌症的预防、诊断和治疗能力。
TCGA主要分为负责测序的GCCs(genome characterization centers)和负责生物信息分析的GDACs(genomedata analysis centers),作为目前最大的癌症基因信息数据库,TCGA的全面不仅仅体现在众多癌症类型上,还体现在多组学数据,包括全基因组、全外显子、转录组测序数据,芯片表达数据,miRNA表达数据,拷贝数变异,甲基化数据,蛋白表达数据,突变数据等,它最大的优势是丰富且规范的临床数据,以及针对每种癌症类型的大样本量。

目前,利用TCGA数据发表的文章越来越多,其中包括癌症亚型鉴定、driver基因挖掘、非编码RNA基因功能研究等热点领域。
表观遗传调控是指不改变DNA序列而对基因表达进行调控的过程。这些特征包括DNA和RNA上的化学修饰, 以及组蛋白翻译后修饰。研究已表明表观遗传在发育、分化过程中起着十分重要的作用,其失调与多种疾病包括癌症的发生发展密切相关。近年来,已有一些靶向表观遗传调控因子的新一代药物陆续处于临床试验中,为新的癌症疗法开发开辟了道路。本项目拟通过整合分析表观遗传调控因子在18种癌症类型中的表达谱、拷贝数变化图谱以及预后图谱,挖掘在特定肿瘤类型中以及在泛癌中呈现最显著异常的表观遗传调控基因,这些基因的异常可能促进了肿瘤的发生发展,有可能作为良好的药物靶标分子。
表观生物通过文献检索,整理了总共493个表观遗传调控因子,包含了159 个组蛋白修饰酶(writer),55 个组蛋白去修饰酶(eraser),116 个组蛋白修饰识别酶(reader),72个染色质重塑因子(chromatin remodellers)58个DNA修饰酶以及33个m6A相关蛋白:

图1 表观遗传调控因子类型
通过整合挖掘TCGA 7332套RNA-seq数据、拷贝数变化数据及临床相关数据,将493个表观遗传调控因子与18种肿瘤进行关联分析(表1),鉴定了异常表达的表观遗传调控因子,发生频繁拷贝数变化的表观遗传调控因子以及显著预后相关的表观遗传调控因子。同时,表观生物评估了与这些表观遗传调控因子共表达的蛋白编码基因,并预测了其可能的生物学功能。这些数据有望为研究癌症相关表观遗传调控因子奠定坚实基础和提升研究效率!
|
类型 |
肿瘤样本数量 |
正常对照数量 |
|
THCA(甲状腺癌) |
504 |
59 |
|
CHOL(胆管癌) |
36 |
9 |
|
UCEC(子宫内膜癌) |
180 |
23 |
|
READ(直肠癌) |
92 |
10 |
|
HNSC(头颈癌) |
518 |
44 |
|
COAD(结肠癌) |
287 |
41 |
|
STAD(胃癌) |
413 |
36 |
|
LIHC(肝癌) |
369 |
50 |
|
KICH(肾嫌色细胞癌) |
66 |
25 |
|
PRAD(前列腺癌) |
494 |
51 |
|
LUAD(肺腺癌) |
513 |
59 |
|
LUSC(肺鳞癌) |
498 |
50 |
|
BRCA(乳腺癌) |
1092 |
113 |
|
KIRP(肾乳头状细胞癌) |
288 |
32 |
|
ESCA(食管癌) |
181 |
13 |
|
KIRC(肾透明细胞癌) |
530 |
72 |
|
BLCA(膀胱癌) |
407 |
19 |
|
GBM(胶质瘤) |
153 |
5 |
表1 肿瘤类型及分析的样本数量
数据应用范围
1. 快速获取在肿瘤中异常表达及存在拷贝数变异的表观遗传调控因子,为验证肿瘤表观遗传调控因子提供理论依据;
2.为相关研究论文的发表提供重要的数据支撑;
3. 可作为申请课题的基础工作,降低前期投入的成本;
4. 为表观遗传调控因子的功能高通量筛选结果提供参照,指导精确锁定肿瘤相关的表观遗传调控因子。
分析内容
1、 差异表达分析
我们通过利用limma包中的voom算法对每种癌症类型中差异表达的表观遗传调控因子进行鉴定,统计显著的表观遗传调控因子被定义为倍数变化大于或等于2倍,假阳性发现率(FDR)小于0.05。以肝癌为例,我们用上述的方法分析了493个表观遗传调控因子在369例肝癌样本与50例癌旁组织样本之间的表达水平,发现183个表观遗传调控因子在癌与癌旁组织之间显著差异表达(见图2)。通过无监督聚类分析,这些基因能够很好的区分肿瘤样本和正常样本。图3显示了肝癌中10个最显著上调的表观遗传调控因子。

图2 热图显示了在肝癌中显著失调的表观遗传调控因子

图3 肝癌中10个最显著上调的表观遗传调控因子
2、 拷贝数变化分析
表观遗传调控因子在癌中异常表达,那是什么因素导致它们发生这种改变呢?我们进一步探索了拷贝数变化对其变化的影响。通过整合分析拷贝数变化和表达数据,我们发现一批显著失调的表观遗传调控因子的DNA拷贝数发生了高频的变化。例如,ENSG00000101189在肝癌中发生频繁的扩增,且明显与它们表达显著正相关(见图4)。
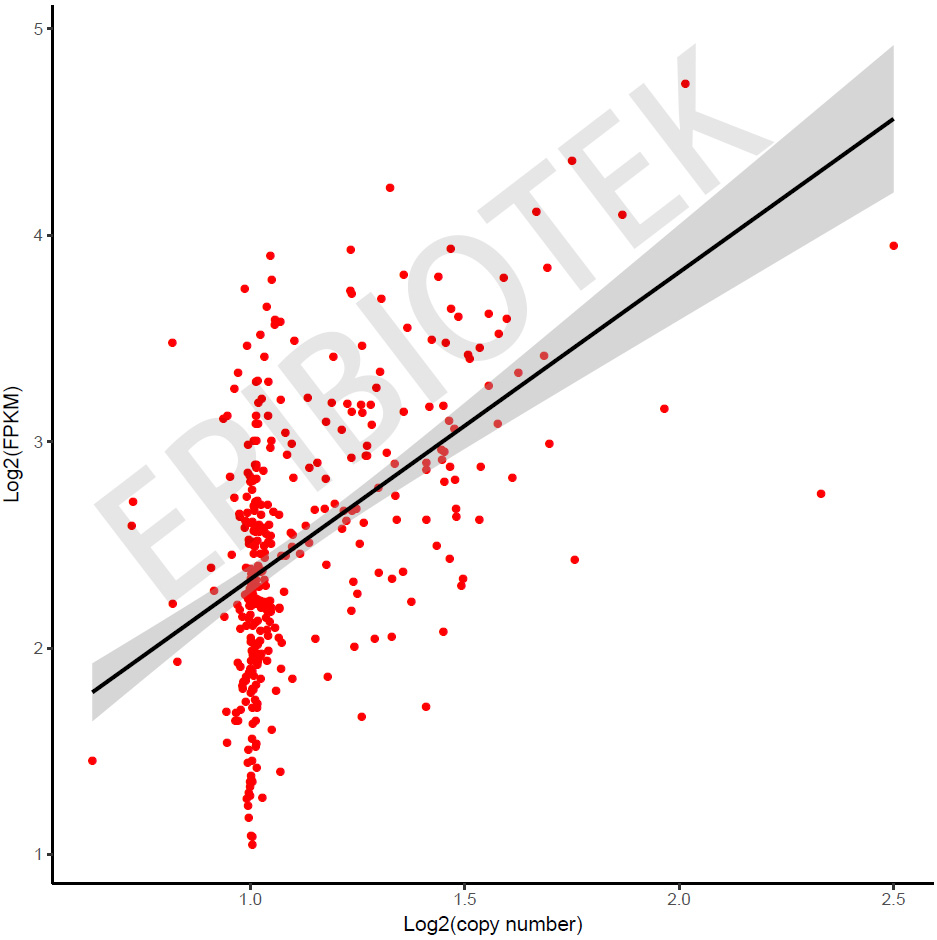
图4 ENSG00000101189在肝癌中扩增,且表达与拷贝数显著正相关
3、 生存分析
我们也基于临床数据,评估了表观遗传调控因子与病人生存之间的关联。基于多因素cox回归模型(以年龄和性别作为协变量),我们鉴定了一批表观遗传调控因子与病人生存显著相关。例如,高表达ENSG00000085999的肝癌病人比那些低表达的病人拥有更短的生存时间(见图5)。
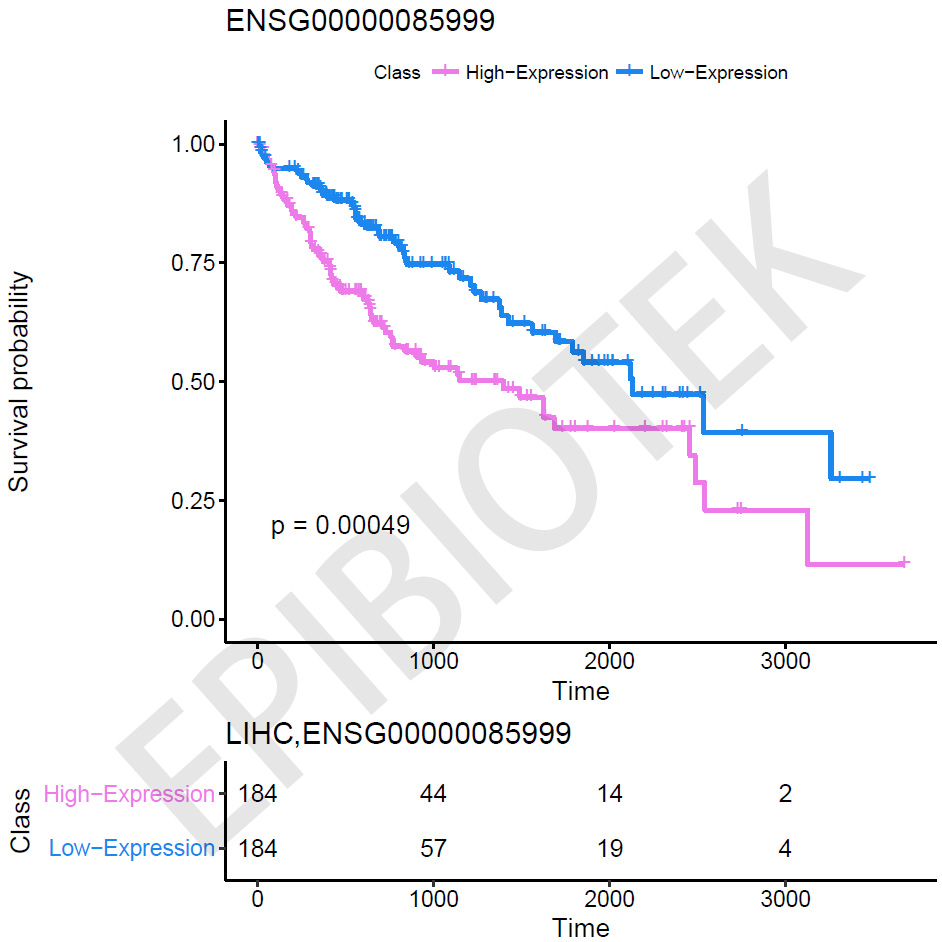
图5 ENSG00000085999的表达与肝癌病人的生存显著相关
4、 鉴定与表观遗传调控因子表达相关的蛋白编码基因
为了研究表观遗传调控因子与蛋白编码基因之间的调控关系,我们分析了表观遗传调控因子与近20000个蛋白编码基因之间的表达相关性。以表观遗传调控因子ENSG00000085999(RAD54L)为例(在肝癌中显著上调),我们鉴定了一组与RAD54L显著相关的蛋白编码基因(FDR<0.05)。图6显示了与PVT1相关的前50个蛋白编码基因,这些基因都与RAD54L高度正相关。基于表达相关性的研究,将有助于我们发现表观遗传调控因子介导的调控关系。
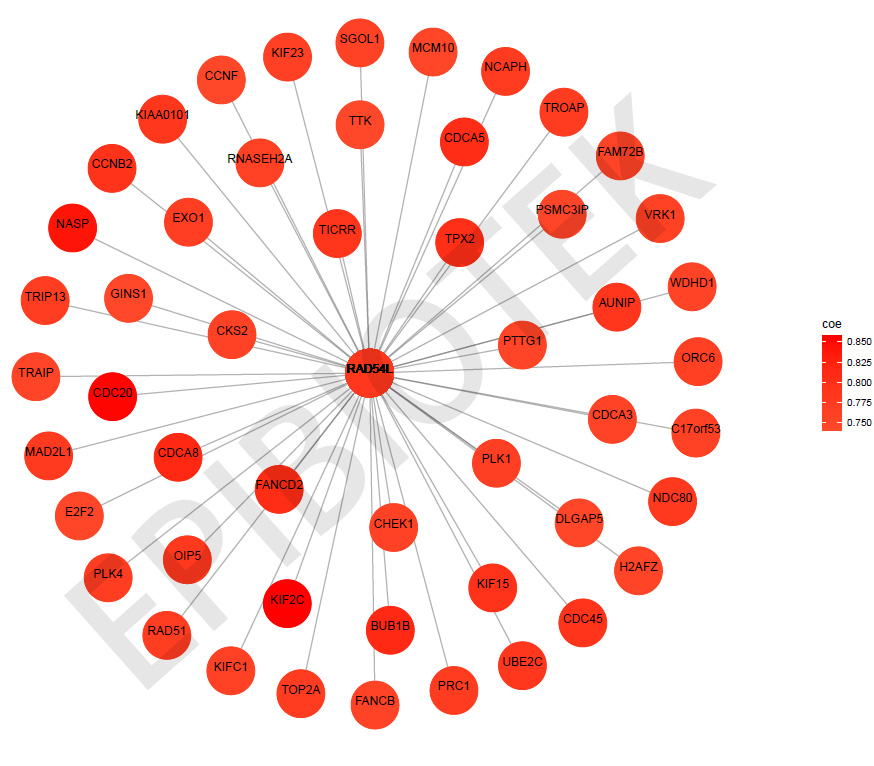
图6 与RAD54L显著相关的前50个蛋白编码基因
5、 表观遗传调控因子功能预测
通过分析与表观遗传调控因子显著相关的蛋白编码基因集,我们能够推断表观遗传调控因子可能的功能。继续以ENSG00000085999为例,通过对与之相关的蛋白编码基因集进行GO分析,我们发现这些基因显著富集在ncRNA加工,组蛋白修饰等生物学过程。KEGG Pathway分析显示这些基因与细胞周期,DNA复制过程显著相关(图7)。
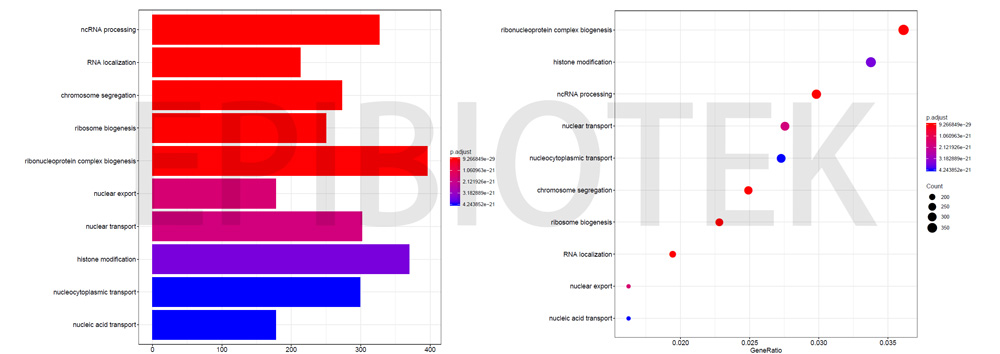
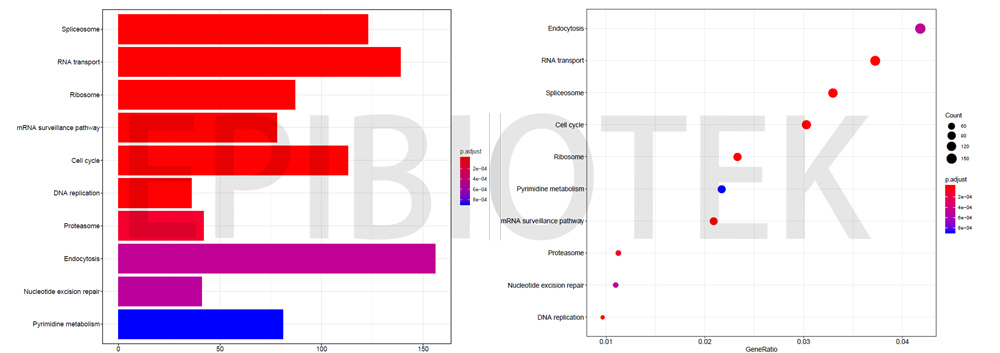
图7 GO和KEGG Pathway分析
注:客户可通过分析结果总表挑选最多10个重点目标的表观遗传调控因子,由表观生物进行差异表达箱线图、表达与拷贝数变化关系图、表达与生存关系图,共表达网络图以及GO和KEGG分析图绘制。(请在原有的结果格式基础上挑出感兴趣的基因返回表观生物,切勿将挑选出的基因再次制定为其它格式返回)
欢迎拨打400-775-0875
或点击本页面右侧【在线咨询】
- - - 推荐阅读 - - -

第二届医学表观遗传学前沿技术培训班圆满落幕!
2019年3月24日,为期三天的第二届医学表观遗传学前沿技术培训班在广州浙江大厦圆满落幕。 培训班现场 本次培训班邀请到了德克萨斯大学
2019-04-01
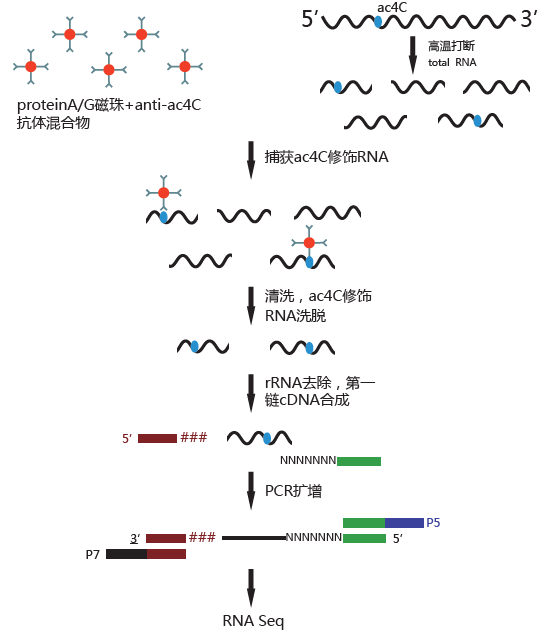
率先推出!RNA乙酰化ac4C测序acRIP-seq
N4-acetylcytidine(ac4C),N4位乙酰胞嘧啶,是真核原核生物中保守的化学修饰,早期研究认为ac4C主要存在tRNA和18SrRNA上[1]。而近期研究显示,mRNA上也存在大量的ac4C,该修饰在促进
2019-03-27
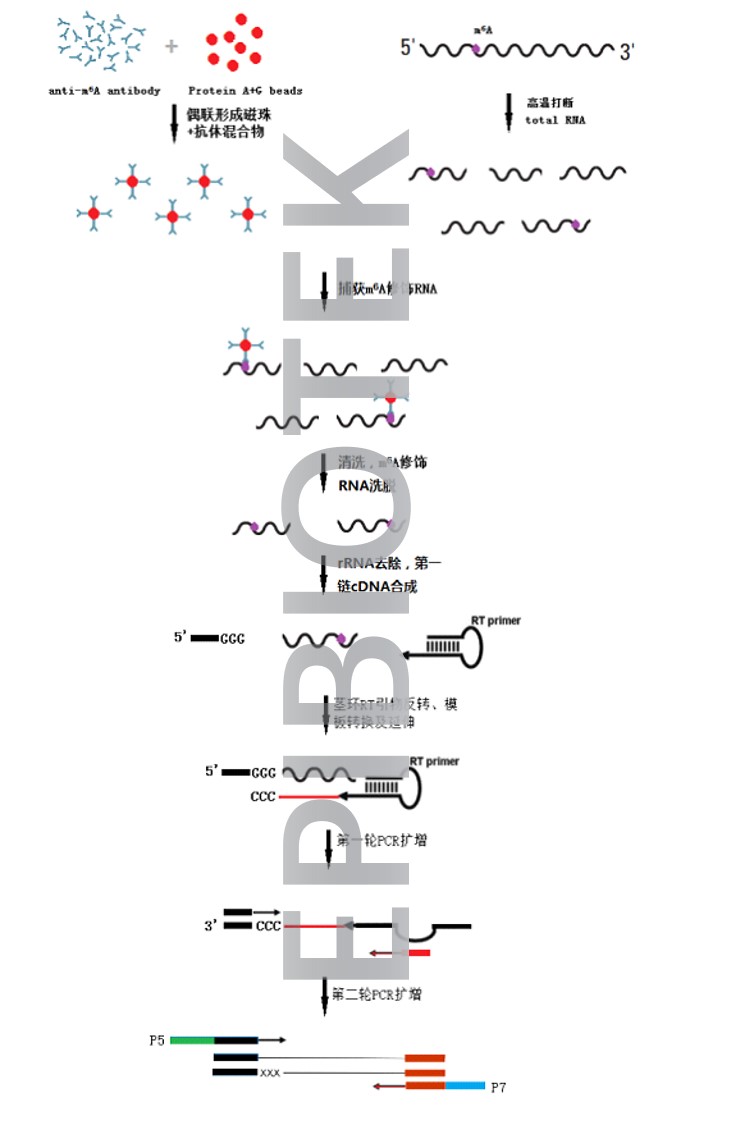
微量RNA甲基化m6A测序(meRIP-seq)技术服务隆重上线
表观生物最新开发微量meRIP-seq技术,通过技术优化,大幅降低meRIP的样本要求量,500ng-20ug 总RNA即可满足实验要求。
2019-01-25
