ONT Direct RNA全长转录组测序
项目简介
ONT Direct RNA全长转录组测序
真核基因通常产生多种RNA异构体(isoform),可以产生功能上不同的蛋白质变体。传统二代测序实际上是一种短读长RNA-seq,难以获得完整的转录本,无法体现异构体的多样性。
表观生物推出ONT Direct RNA全长转录组测序服务,基于Oxford Nanopore公司的纳米孔测序平台,直接对RNA进行测序,具有长读长的优势,提高对异构体的检测,还能直接检测m6A、假尿嘧啶pseudoU等核酸修饰,在可变剪切研究方面具有独特的优势。一次测序,即可满足多项表观遗传转录组研究需求,大大提升研究的高度、深度、准确性、效率,尤其适用于多组学分子机制及珍贵的临床样本研究。
应用与优势
技术应用
1. 对单个RNA分子进行单分子水平的全长测序
2. 识别更长的转录本,研究转录异构体
3. 绘制转录组上的m6A修饰图谱,分辨率可达到单位点
4. 检测RNA可变剪切的情况
技术优势
1. 作为长读长测序技术,能减少短读长测序打断后重新拼接带来的歧义,结果准确度更高、更可信;
2. 能够直接对RNA进行测序,避免逆转录和PCR扩增导致的RNA修饰信息缺失;
3. 可以直接检测m6A等碱基修饰,配合优化的算法,获得无损的m6A单碱基分辨率图谱;
4. 一次测序,多套数据:转录组图谱、m6A修饰图谱、异构体、可变剪切情况,节省珍贵的临床样本,节省样本准备时间和精力,且保持了样本的统一性。
送样要求
样本物种: 仅限人、大小鼠,其他物种需评估
>20ug
总RNA
1x10⁷
细胞数量
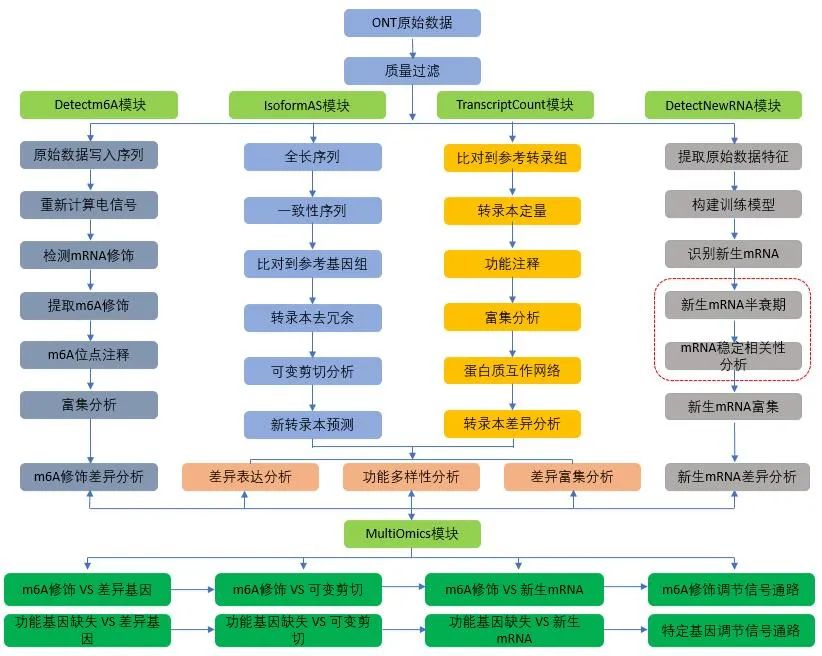
分析流程
分析内容
一、isoform分析
1. 可变剪切分析
2. 融合基因鉴定
3. SSR分析
4. LncRNA分析
5. PolyA分析
二、表达定量
1. 转录本定量
2. 转录本差异分析
3. 富集分析
4. 蛋白质互作网络
三、甲基化修饰分析
1. m6A位点注释
2. 甲基化修饰富集分析
3. m6A差异分析
四、isoform联合定量表达分析
1. AltTP分析
2. 功能多样性分析(FDA)
3. 差异富集分析
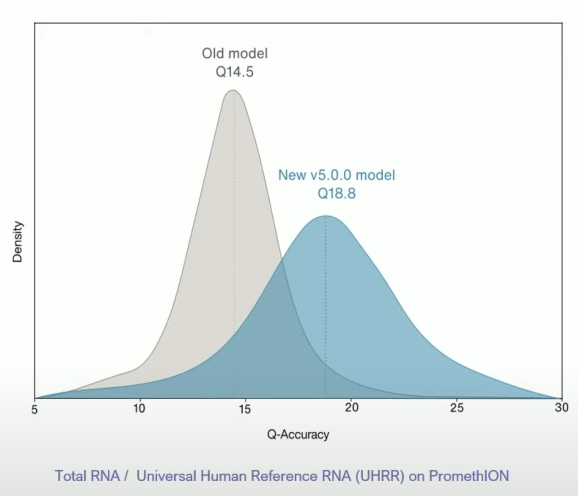
图1. 不同basecalling模式下Q值的比较。单分子测序原始reads的中位准确率高达98.8%
结果示例
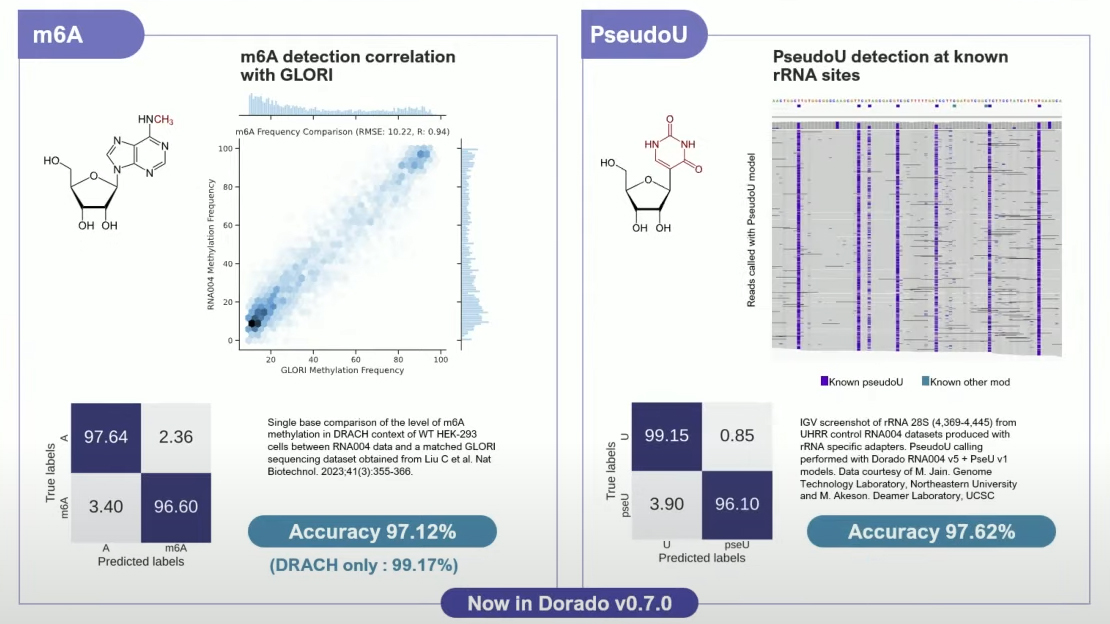
图2. 同时分析m6A和pseudoU,识别准确率分别为97.1%和97.6%
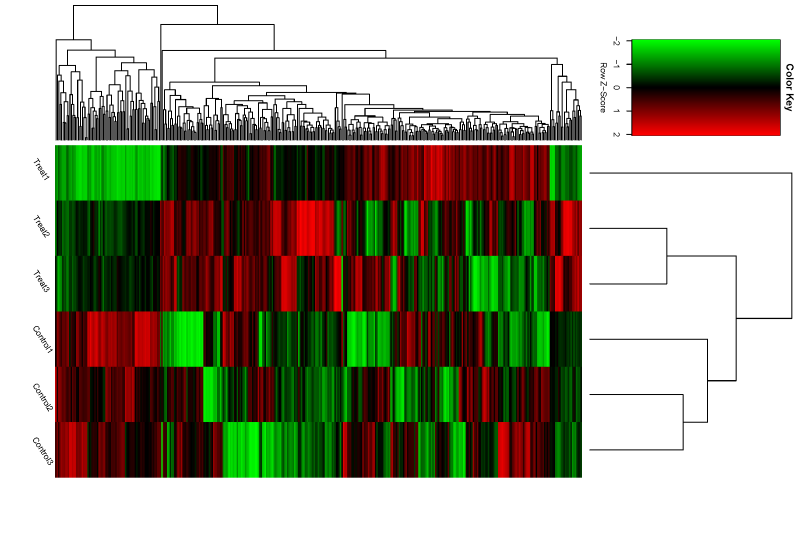
图3. isoform差异热图
表观生物实测数据
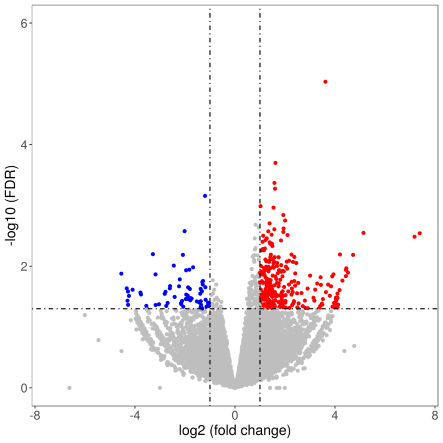
图4. isoform显著差异火山图
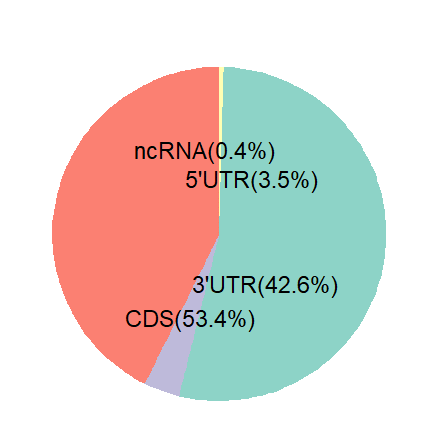
图5. m6A在RNA结构上的分布饼图
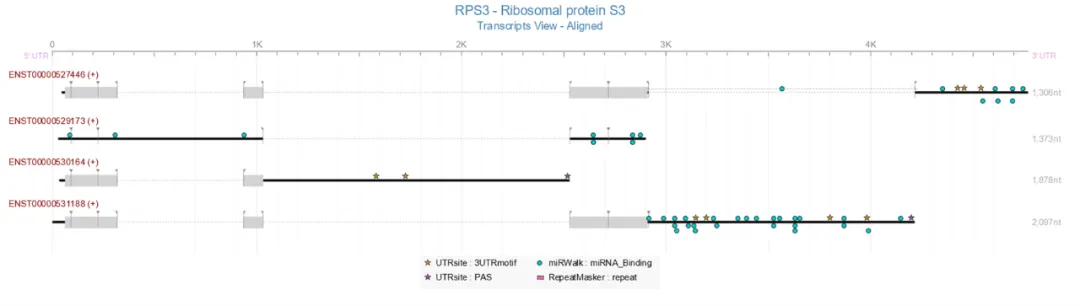
图6. 全长转录本水平-差异分析
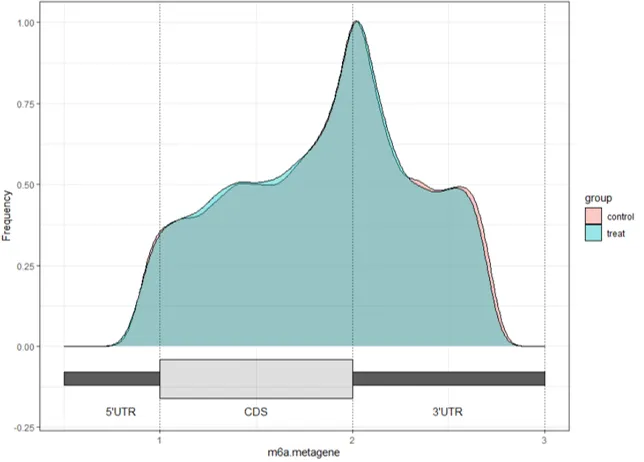
图7. 甲基化修饰-m6A修饰位点分布
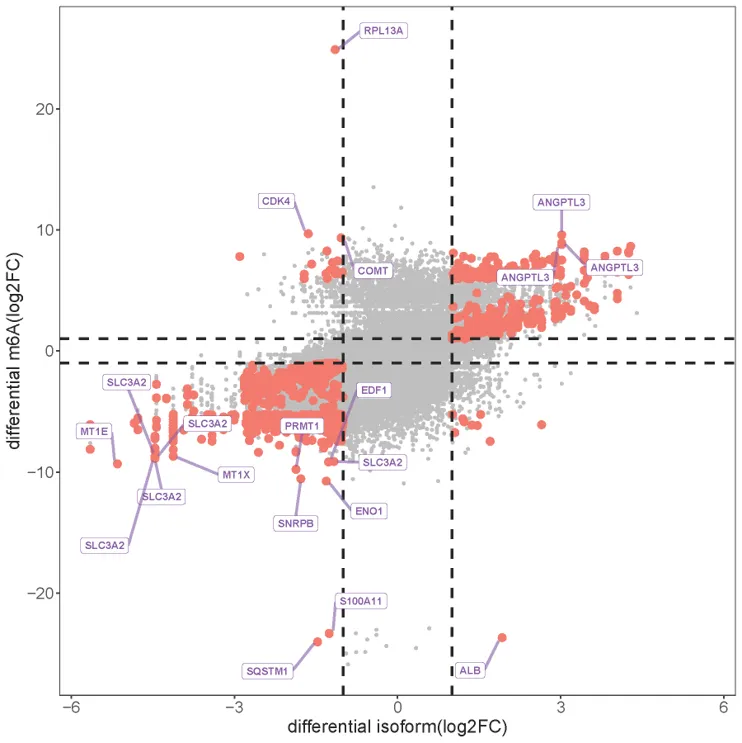
图8. 联合分析-多维度和IPA结果
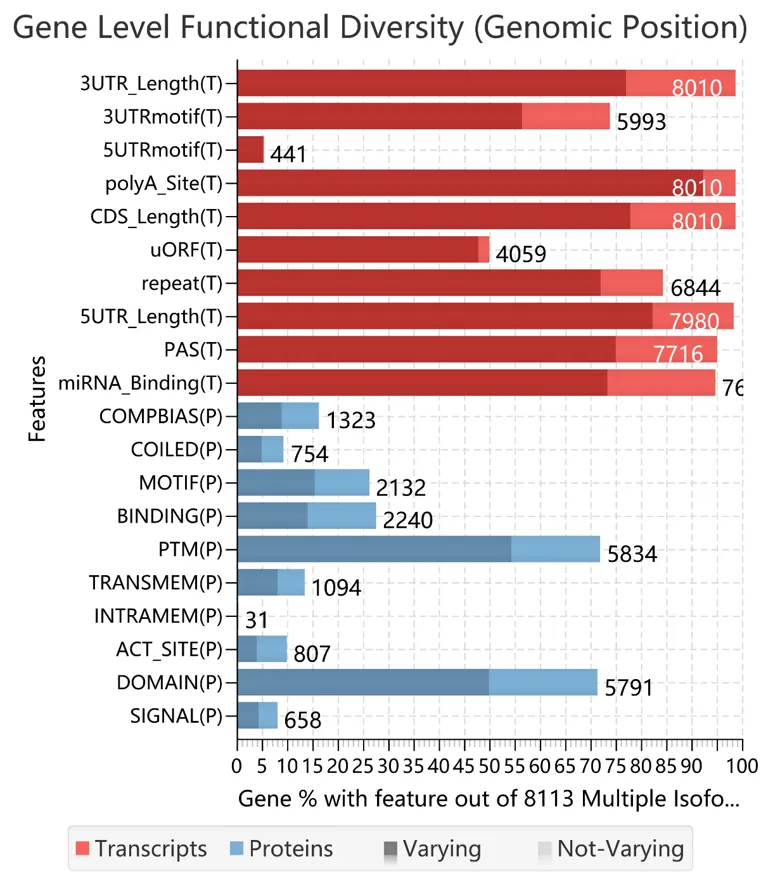
图9. 全长转录本-整体变化

图10. 全长转录本-DIU分析
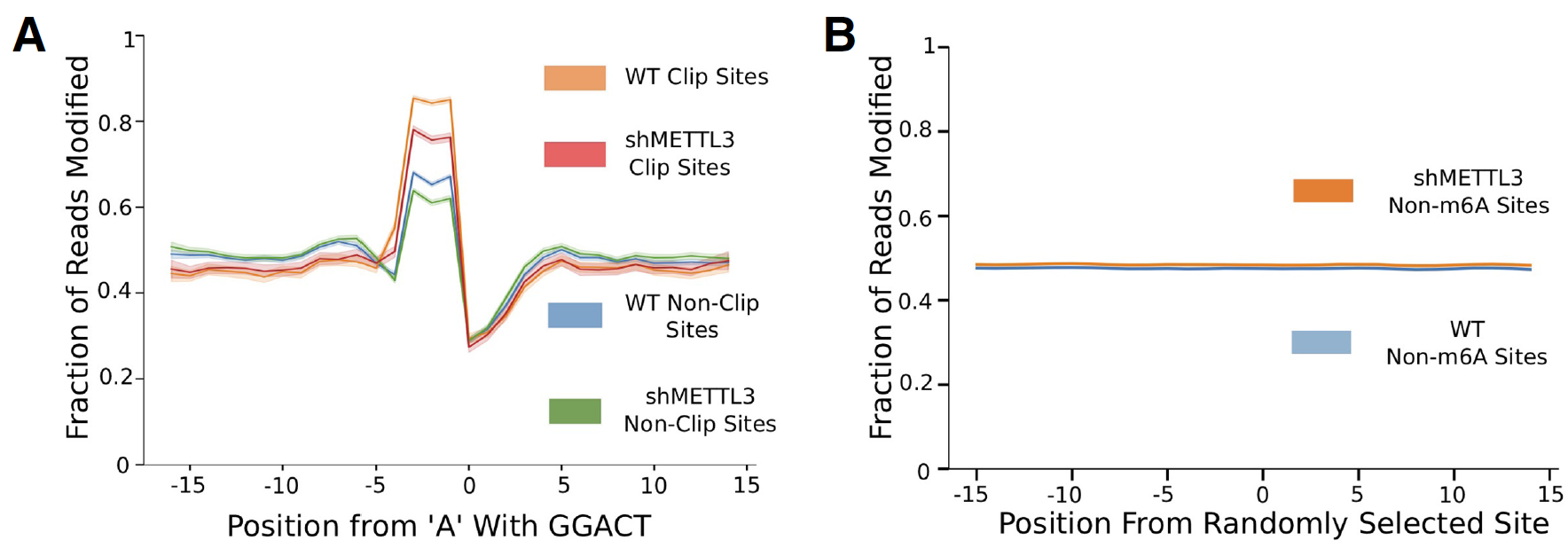
纳米孔测序能从DRACH motif中检测出m6A修饰
案例解读
RNA直接测序应用于鉴定内源转录本异构体的m6A修饰
为了将RNA直接测序应用于单位点分辨率的RNA修饰从头测序,研究员研发了利用纳米孔RNA直接测序的m6A鉴定软件(MINES),对HEK293T细胞转录本进行了分析,鉴定超过13,000个之前未注释的DRACH位点的m6A甲基化状态;还在人乳腺上皮细胞(包括异构体)中鉴定得40,000个位点,这些位点分别对m6A writer、METTL3、eraser、ALKBH5敏感。以上数据表明,纳米孔RNA直接测序鉴定m6A修饰的效果优异。
原文:DANIEL A. LORENZ, et al. Direct RNA sequencing enablesm6A detection in endogenous transcript isoforms at base-specific resolution. RNA(2020) 26:19–28.
