CUT&Tag:蛋白质-DNA互作研究技术,鉴定表观基因组修饰图谱

技术应用
Cleavage Under Target & Tagmentation (CUT&Tag) 是近年来新兴的一种染色质DNA结合图谱测序研究方法。该方法通过Protein A/G融合的Tn5转座酶与抗体结合,使Tn5转座酶被募集在靶标位点附近,从而在靶标位点引发DNA转座反应,在将染色质DNA切断的同时,引入二代测序所需的接头序列。CUT&Tag技术相比于经典的ChIP-seq技术,具有不需要超声打断,信噪比高,所需细胞量少的优势。此外,相比于另一种新技术Cleavage Under Target And Release Using Nuclease (CUT&RUN),CUT&Tag不需要传统的连接法添加测序接头,操作简单、实验周期更短。因此,CUT&Tag在组蛋白修饰、转录因子的染色质结合状态的研究中得到了广泛的应用。
特大福利
表观生物CUT&Tag技术服务
免费提供多种组蛋白修饰抗体
以及转录因子抗体
欢迎咨询!
技术应用
H3K27ac:绘制增强子图谱,鉴定超级增强子目录
H3K4me3:检测转录起始位点附近激活的启动子区域
组蛋白乳酸化修饰:在基因启动子上高度富集,与Warburg效应相关的血管生成、缺氧、巨噬细胞极化过程相关基因的活跃表达有关。
组蛋白巴豆酰化修饰:与活跃转录和基因激活密切相关,调控着基因表达及配子成熟等生物学过程。此外,组蛋白巴豆酰化修饰的蛋白参与了RNA剪接、蛋白质合成降解、DNA复制和修复、内吞作用、细胞间连接等多种生物学过程。
DNA G4链体:通常位于基因启动子区域、5'非翻译区和染色体端粒,能够影响这些区域的DNA复制、转录和稳定性。例如,G4结构的形成可以作为一种调节机制,通过阻碍或促进转录因子的结合来调节特定基因的表达,与多种疾病的发展有关,如癌症、神经退行性疾病等。
更多转录因子:挖掘蛋白质-DNA相互作用
技术优势
1. 样本量仅需20万个细胞
2. 操作便捷,一管完成所有步骤
3. 信噪比高(背景低),重复性好
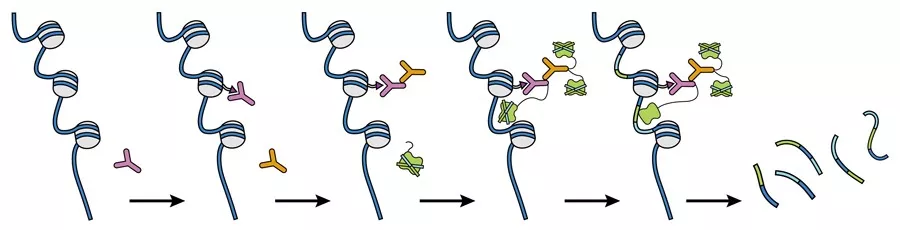
图1. CUT&Tag技术原理图
技术原理
将特异的抗体(如H3K27ac)与染色质蛋白原位结合,然后锚定到A-Tn5转座融合蛋白。Tn5转座酶活性被激活,仅将抗体靶蛋白结合的染色质片段化,高效产生高分辨率、低背景的片段文库。
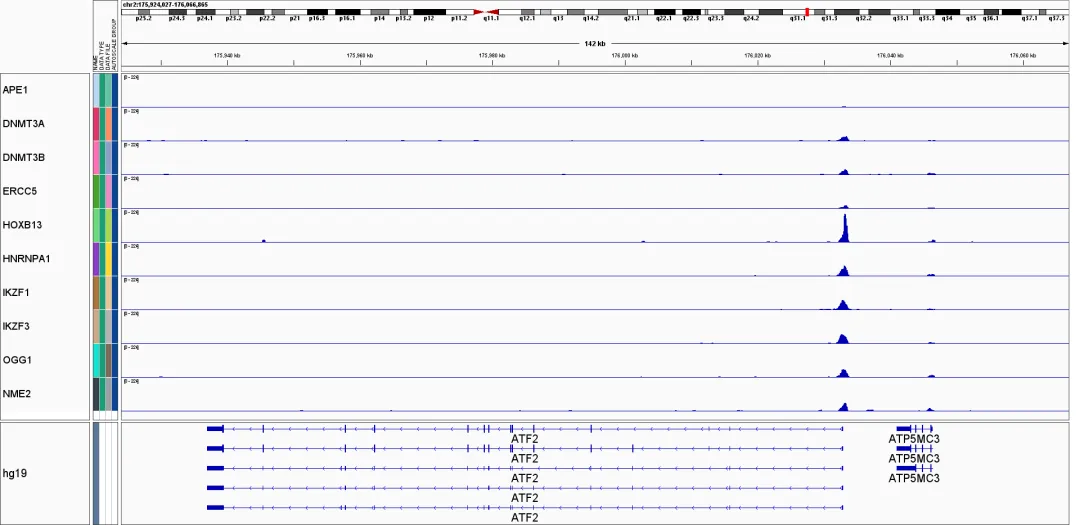
图2. IGV峰图
表观生物实测数据
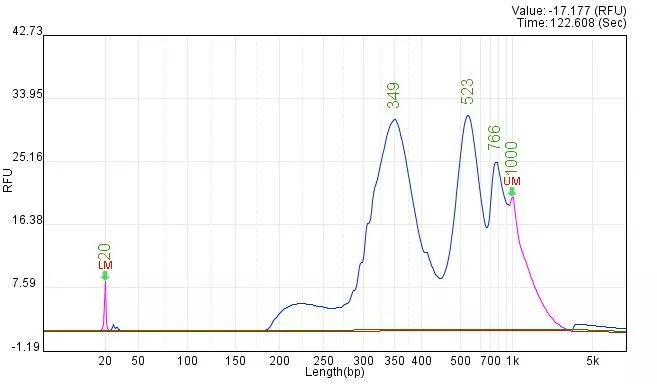
图3. CUT&Tag文库质控图
![图4. 表观生物利用CUT&Tag检测组蛋白修饰H3K27me3的读长覆盖图,显示CUT&Tag H3K27me3 peaks(第二行),与文献数据对比(第一行)[1]](http://openapi.whaleng.com/file-server/ep/2024/06/01ac71642a334d74b46eb8df6a1ba376.png)
图4. 表观生物利用CUT&Tag检测组蛋白修饰H3K27me3的读长覆盖图,显示CUT&Tag H3K27me3 peaks(第二行),与文献数据对比(第一行)[1]
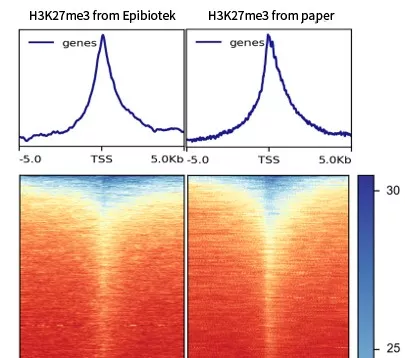
图5. 表观生物利用CUT&Tag检测H3K27me3的TSS分布热图(左列),与文献数据对比(右列)[1]
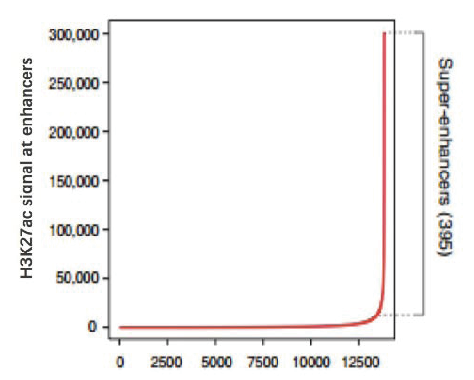
图6. 普通增强子与超级增强子鉴定分析
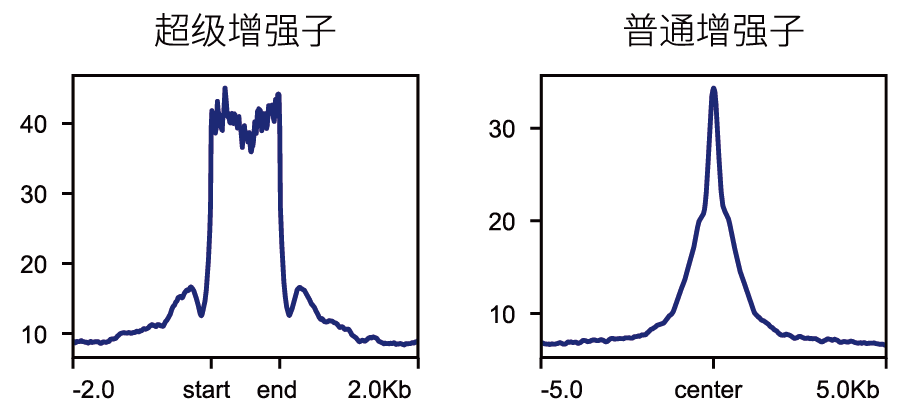
图7. 普通增强子与超级增强子信号富集图
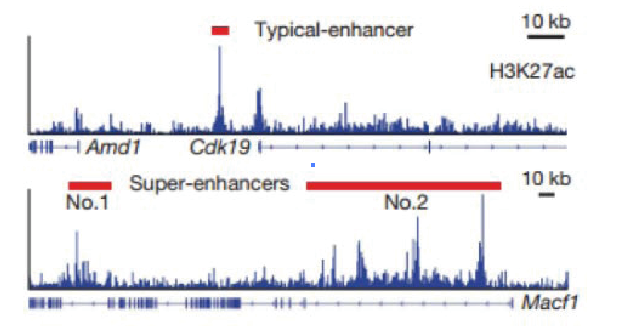
图8. 超级增强子鉴定及数量分析图绘制
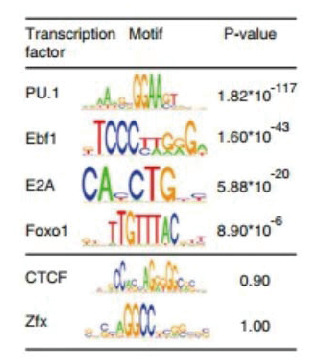
图9. 超级增强子结合转录因子预测
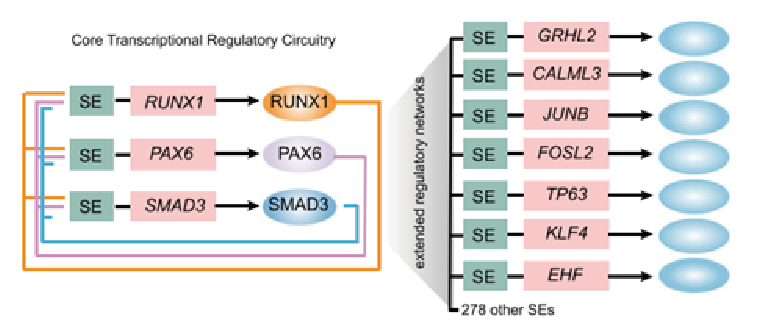
图10. 核心转录因子调控回路(CRC)
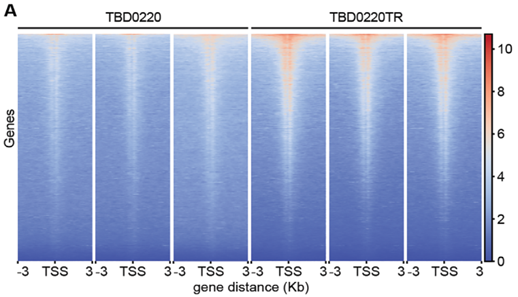
图11. 热图显示H3K9la CUT&Tag测序信号在TBD0220TR细胞有明显的富集
客户文章
Adv Sci:组蛋白乳酸化修饰在胶质母细胞瘤耐药中的作用机制研究[2]
此研究通过联合CUT&Tag、SLAM-seq和RNA-seq等技术,研究了组蛋白H3K9la乳酸化在GBM TMZ耐药中的作用,详细解析了下游调控机制,为临床GBM的治疗提供了潜在的联合治疗策略。
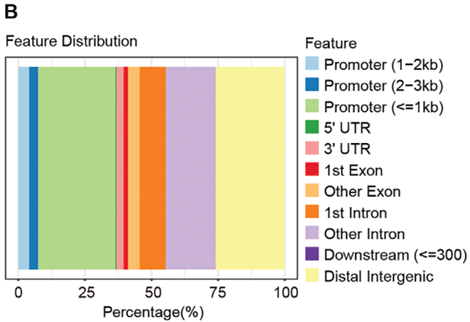
图12. TBD0220TR细胞中上调的H3K9la富集峰大部分位于启动子区
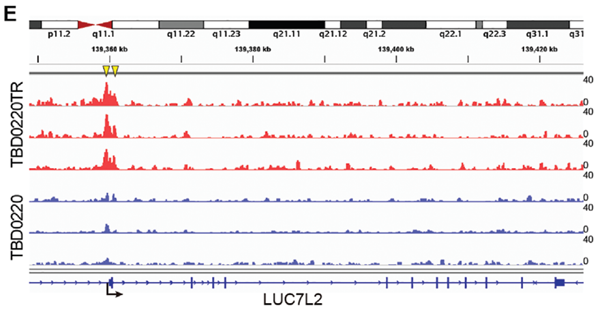
图13. IGV图显示LUC7L2启动子上的H3K9la峰
Sci Transl Med:超级增强子图谱揭示慢性粒细胞白血病的潜在靶向治疗策略[3]
此研究首次绘制了白血病干细胞的超级增强子图谱,明确白血病干细胞的功能维持高度依赖超级增强子驱动的基因转录,干预超级增强子相关的基因转录成瘾杀伤白血病干细胞。研究者鉴定出XBP1是白血病干细胞命运决定的关键超级增强子相关基因。这项研究从超级增强子的角度阐明白血病干细胞自我更新调控的新机制,丰富了癌基因成瘾性学说,为靶向清除白血病干细胞提供了一种新的治疗策略和小分子化合物。
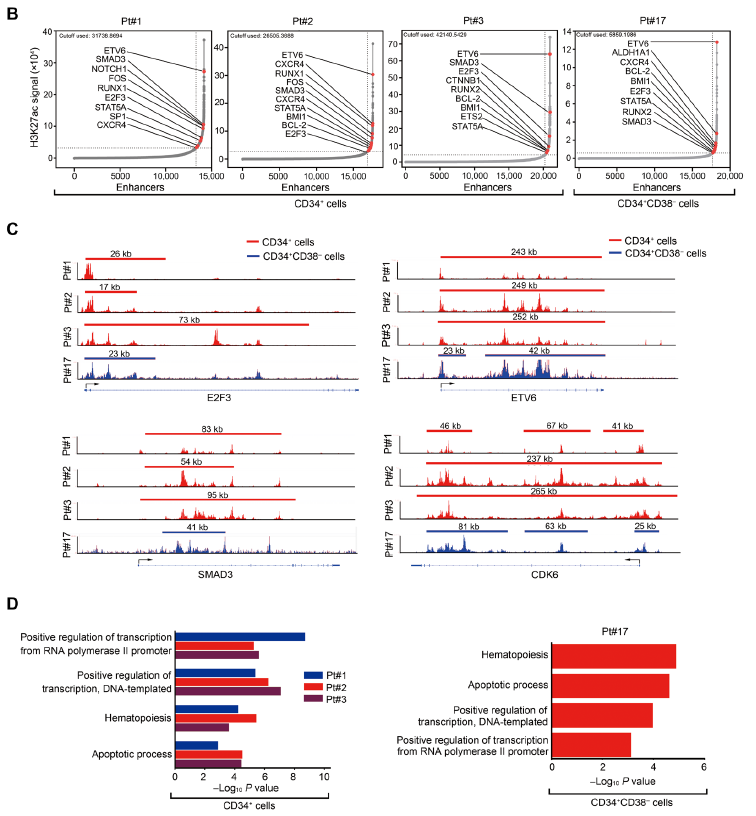
图14.原代CML CD34+ 和CD34+CD38−细胞的超级增强子鉴定分析
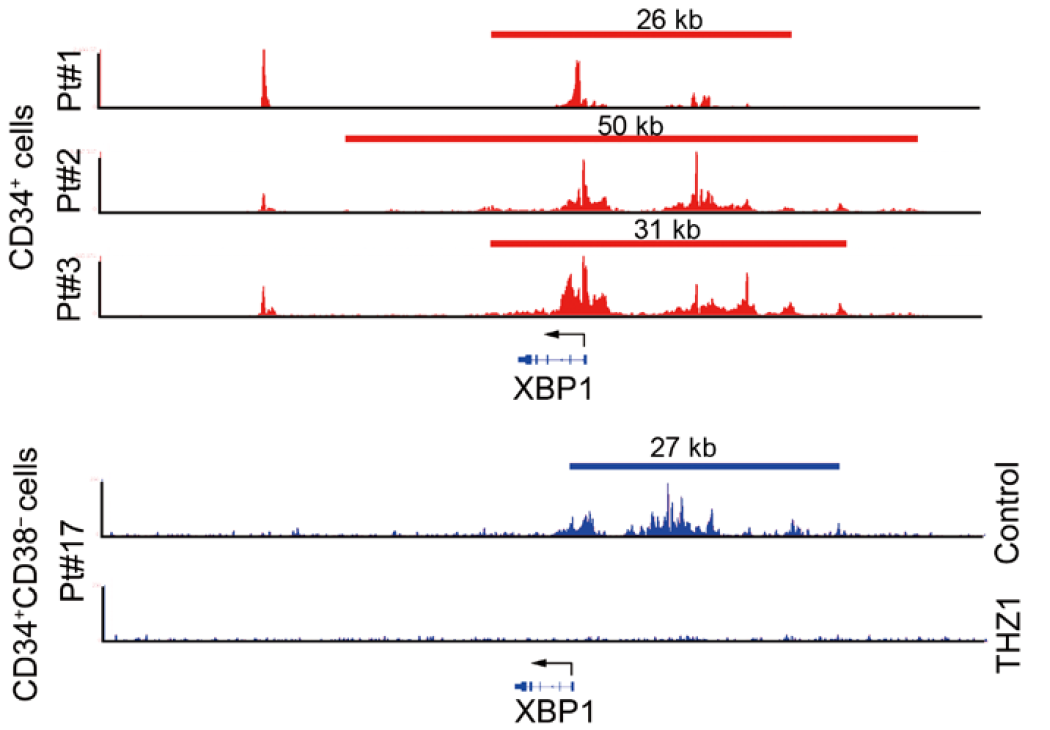
图15. ChIP-seq结果显示H3K27ac与 XBP1基因的结合位点
参考文献
[1] Kaya-Okur HS, Wu SJ, Codomo CA, et al. CUT&Tag for efficient epigenomic profiling of small samples and single cells. Nat Commun. 2019 Apr 29;10(1):1930.
[2] Yue Q, Wang Z, Shen Y, Lan Y, Zhong X, Luo X, Yang T, Zhang M, Zuo B, Zeng T, Lu J, Wang Y, Liu B, Guo H. Histone H3K9 Lactylation Confers Temozolomide Resistance in Glioblastoma via LUC7L2-Mediated MLH1 Intron Retention. Adv Sci (Weinh). 2024 May;11(19):e2309290. (客户文章)
[3] Zhou J, Wang S, Nie D, et al. Super-enhancer landscape reveals leukemia stem cell reliance on X-box binding protein 1 as a therapeutic vulnerability. Sci Transl Med. 2021 Sep 22;13(612):eabh3462. (客户文章)
[4] Huang Y, Wang X, Hu R, Pan G, Lin X. SOX2 regulates paclitaxel resistance of A549 non‑small cell lung cancer cells via promoting transcription of ClC‑3. Oncol Rep. 2022 Oct;48(4):181. (客户文章)
