Hi-C
项目简介
Hi-C
Hi-C(High-throughput chromosome conformation capture),即高通量染色体捕获技术,可在全基因组范围研
究 DNA 的相互作用,描绘高分辨率的染色质三维结构图谱,得到 A/B compartment、拓扑相关结构域(TAD)、染色质
环(loop)三个层级的信息。作为表观遗传学科研服务的国内先驱,表观生物可为客户提供 Hi-C 技术与表观多组学研究
手段联合分析的整体研究思路,从三维表观遗传组学层面深入研究疾病发生发展的机制。
应用与优势
技术应用
1. 研究染色质片段之间的相互作用
2. 建立基因组三维折叠模型
3. 适用于基因组组装
4. 与 RNA-seq 联合分析,研究染色质三维构象对
RNA 表达的调控
5. 与 ChIP-seq 联合分析,解析调控元件、转录因子
与染色质相互作用的关系
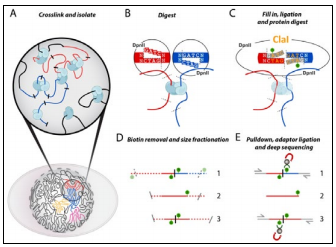
图 1-3. Hi-C 技术原理
技术原理
A, 甲醛固定细胞;B. DpnII 消化 DNA,识别 GATC,生成 5'-GATC 粘性末端;C. 用 dNTPs 和生物素 -14-dATP 填平 5'
粘性末端,成为平端。连接平端,生成一个新的限制性酶切位点(Clal),可用于评估填平效率。连接之后,交联逆转,
去除 DNA 中的蛋白质;D. 去除未连接端的生物素。DNA 被切成 200~300bp 的片段,适用于双端测序;E. 用链霉亲和素
磁珠富集连接产物,再加接头。
送样要求
细胞,≥ 5×106 个细胞 / 样本
样本类型
≥ 100mg/ 样本
组织
需要先用甲醛交联固定。
分析内容
1. 数据过滤
1.1 原始数据
1.2 数据过滤
1.3 测序数据质量分布
1.4 测序数据碱基分布
2. 基因组比对分析
3. 比对到酶切片段
4. 顺反相互作用比例
5. 染色体相互作用
5.1 染色体间相互作用
5.2 染色体内的相互作用
6. 结构分析
6.1 A/B compartments
6.2 TAD calling
6.3 Loop 鉴定及差异分析
6.4 三维建模
7 结构分析
7.1 A/B compartments
7.2 TAD calling
7.3 Loop 鉴定及差异分析
7.4 三维建模
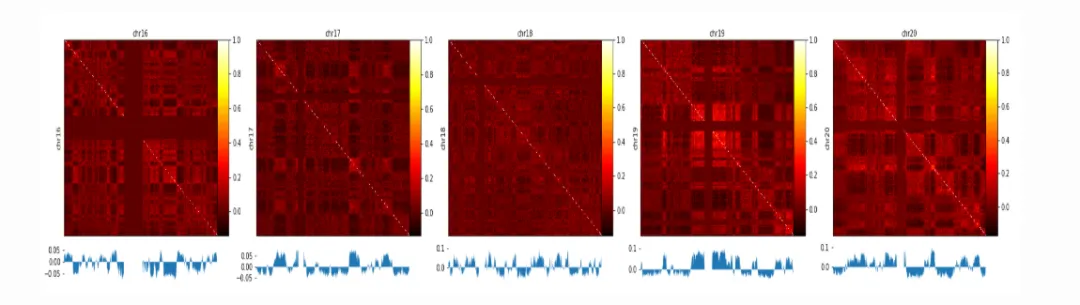
图2. A/B compartment分析
分析示例

图3. TAD鉴定
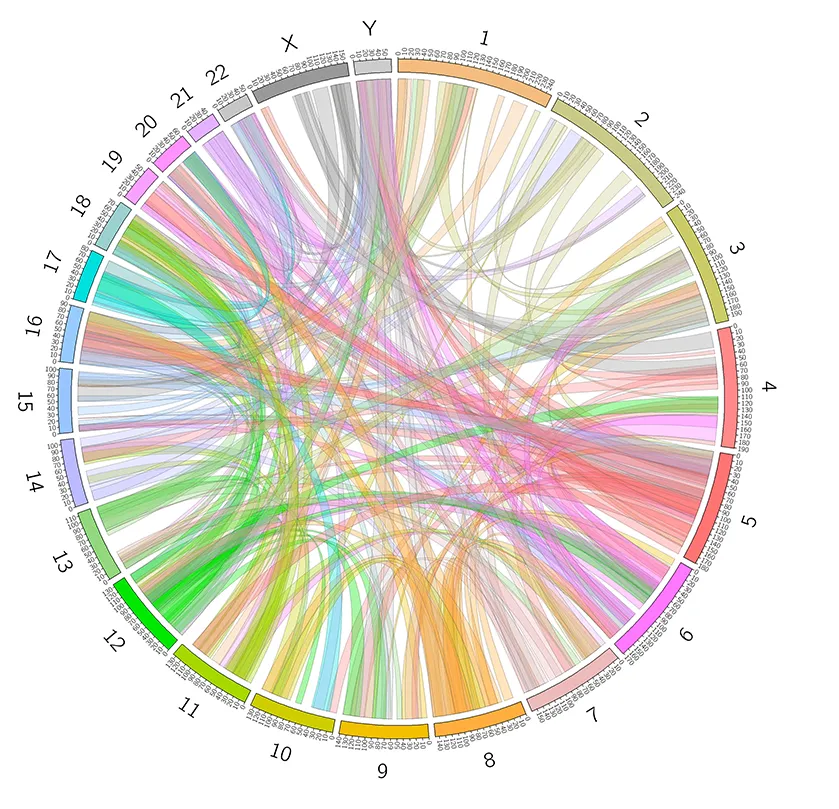
图5. Loop鉴定
图6. Loop差异分析
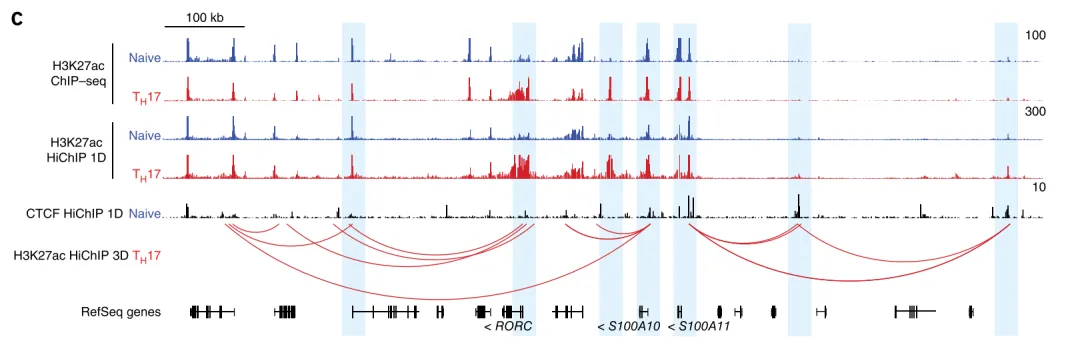
图8. SNP与Loop关联分析(感兴趣基因绘图)
参考案例
Hnisz D, et al. Cell, 2013. Hi-C 分析揭示黏连蛋白对浆细胞瘤发展的影响 [1]
目前研究认为黏连蛋白挤压对哺乳动物基因组的架构建立有着重要的影响,但是我们对它的功能影响和能量特性都
是未知的。研究者利用深度极高的 Hi-C,表明 loop 结构域形成的过程中需要黏连蛋白 ATP 酶,并在 loop 锚定点找到结
构性的“stripes”,它们与整个结构域有着高频率的相互作用,促进超级增强子与启动子的相互作用。在浆细胞瘤中,
stripes 可以调控 lgh 易位的致癌基因。研究者猜想高等生物利用黏连蛋白挤压,增强基因转录和重组,与肿瘤发展有关。
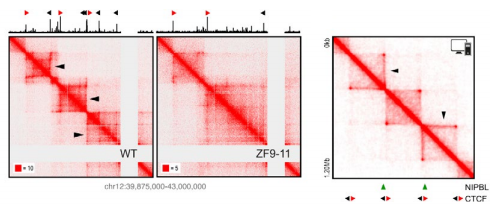
图 9 左图 . Hi-C 矩阵表明 ZF9-11 细胞 stripe 锚定位点失去 stripes 之后,不能募集 CTCF;右图 . 分子动态刺激的 Hi-C 数据,显示黏连
蛋白在 CTCF 锚定位点的附近。
![Nat Commun:整合Hi-C和短读长、长度长基因组测序,揭示种系重组基因组的结构[2]](http://openapi.whaleng.com/file-server/ep/2024/11/dd06a04babff41459ec7e35585d841c1.png)
图10. 对于大多数复杂的染色体破碎情况,因为无法识别所有新的邻接所以重建图并不完整,会停止在染色体支架不完整的地方。通过Hi-C,研究者用断点定义2D网格,并在其内搜索支架之间共同的异位Hi-C接触,重建了衍生染色体的结构,弥补了二代、三代全基因组技术的缺陷
Nat Commun:整合Hi-C和短读长、长度长基因组测序,揭示种系重组基因组的结构[2]
此研究利用Hi-C、全基因组测序、转录组测序,多组学联合揭示了人基因重排3D染色质图谱,表明基因重排事件对TAD改变,以及基因组的大规模染色体重排的整体健壮性(robustness)。
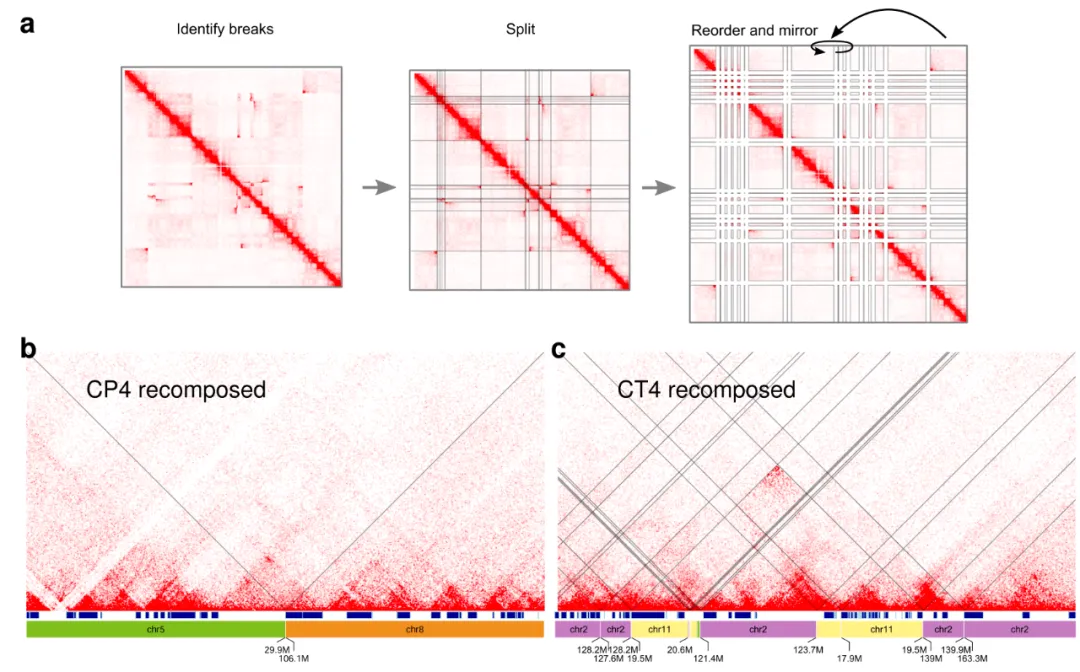
图11. Hi-C技术揭示重排染色体的染色质结构(a图),显示了大量的TAD融合和循环(b、c图)。
![Cell Death Differ:hnRNPK调控染色质三维结构以维持小鼠胚胎肢芽发育[3]](http://openapi.whaleng.com/file-server/ep/2024/11/8761b26a7aa8411e8845c63ad92599d8.webp)
这项研究揭示了异质核核糖核蛋白K(hnRNPK)在小鼠胚胎肢芽发育中的关键作用。研究人员利用Hi-C、RNA-seq、ATAC-seq、CUT&Tag和CUT&RUN技术,发现hnRNPK缺失导致小鼠胚胎前肢缺失、后肢畸形。机制研究表明,hnRNPK作为转录激活因子,调控肢芽发育关键基因的表达。此外,hnRNPK还与染色质架构蛋白CTCF相互作用,共同维持染色质三维结构的稳定。hnRNPK缺失导致CTCF在TAD边界处的结合强度降低,进而削弱启动子和增强子的相互作用,最终导致肢芽发育关键基因表达下调。
Cell Death Differ:hnRNPK调控染色质三维结构以维持小鼠胚胎肢芽发育[3]
图12. Shh-TAD在野生型和hnRNPK敲除小鼠胚胎肢芽细胞中的Hi-C交互热图,绿色方框突出显示了Shh启动子和ZRS增强子之间相互作用的减弱
参考文献
[1] Laura Vian , Aleksandra Pękowska , Suhas S P Rao, et al. The Energetics and Physiological Impact of Cohesin Extrusion. Cell. 2018 May 17;173(5):1165-1178.e20.
[2] Schöpflin R, Melo US, Moeinzadeh H, et al. Integration of Hi-C with short and long-read genome sequencing reveals the structure of germline rearranged genomes. Nat Commun. 2022 Oct 29;13(1):6470.
[3] Chen Y, Zhou T, Liao Z, et al. Hnrnpk is essential for embryonic limb bud development as a transcription activator and a collaborator of insulator protein Ctcf. Cell Death Differ. 2023;30(10):2293-2308.
