CUT & RUN 技术服务
项目简介
CUT&RUN技术服务
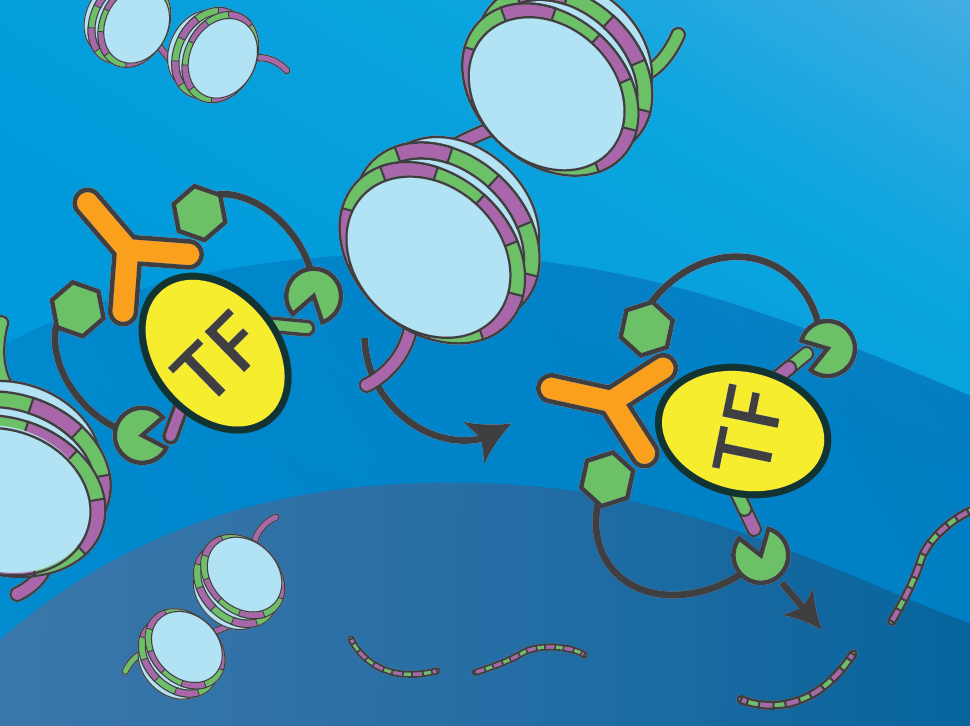
CUT & RUN(cleavage under targets and release using nuclease),即核酸酶靶向切割和释放技术,是一项用于在细胞天然染色质环境下检测蛋白-DNA复合体的新方法,与传统的ChIP-Seq研究方法相比,该技术无需甲醛进行交联剂免疫沉淀等操作,因此具有快速、所需低细胞量,背景信号低、重复性好等优点,可用于单细胞水平研究。CUT & RUN可作为表观遗传、基因调控等领域的研究具有革命性意义。
技术原理
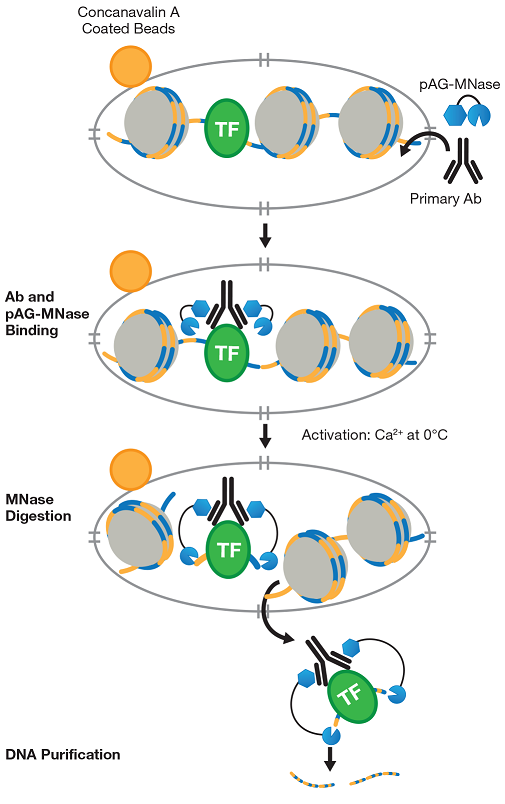
无需用甲醛进行交联剂免疫沉淀,通过针对靶蛋白(如转录因子、染色质重塑蛋白)特异性抗体和特异结合免疫球蛋白G(Protein A)的介导,使与Protein A融合的pAG-微球菌核酸酶(pAG-MNase)切割并释放靶蛋白结合的序列,获得目的DNA片段,并进一步建库测序。
应用与特点
技术应用
1. 挖掘蛋白质—DNA相互作用
2. 检测基因组上的组蛋白修饰,鉴定超级增强子
3. 检测基因组的开放性、转录因子结合图谱,鉴定超级增强子
4. 绘制核小体、RNAPII等蛋白的染色质图谱
技术特点
1. 样本量仅需20万个细胞(组蛋白)
2. 数据信噪比高,背景低,数据重复性好;
3. 操作简便,实验操作时间短,仅1天。
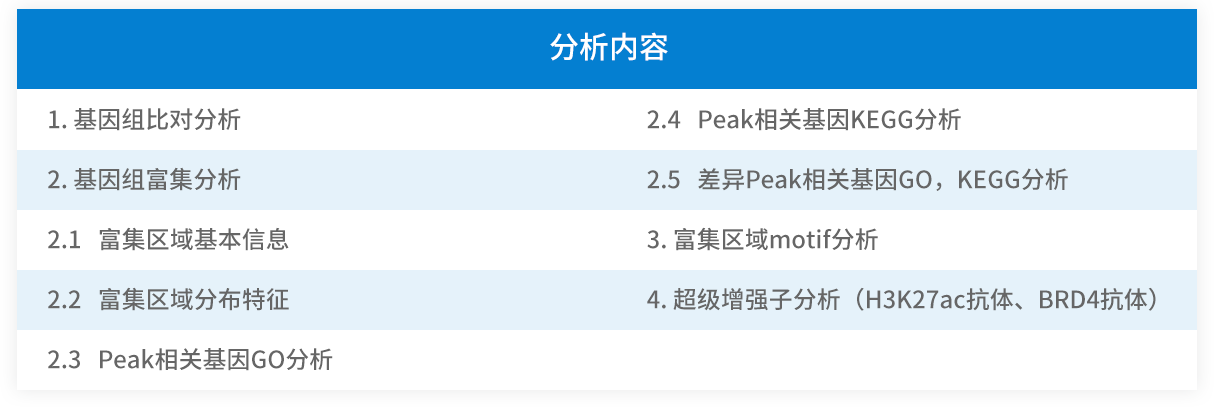
分析内容
送样要求
活细胞
样本类型
5 x10^5
细胞数量
人、大小鼠
样本物种
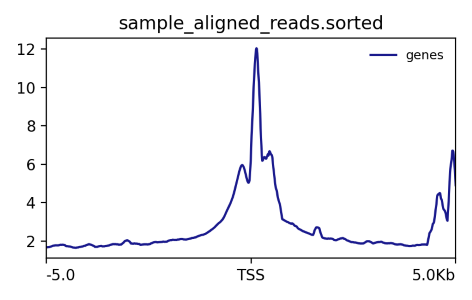
图1. reads分布在TSS附近。*注释:reads分布在TSS附近
表观生物实测数据

图2:TSS附近的heatmap图。*注释:由热图可见,TSS附近reads富集程度
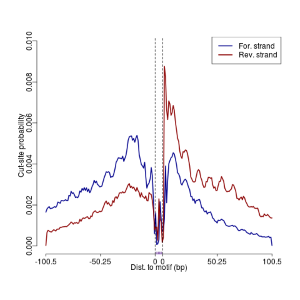
图3:footprinting图 。*注释:motif的footprinting可查看motif在各个位置上的切割频率
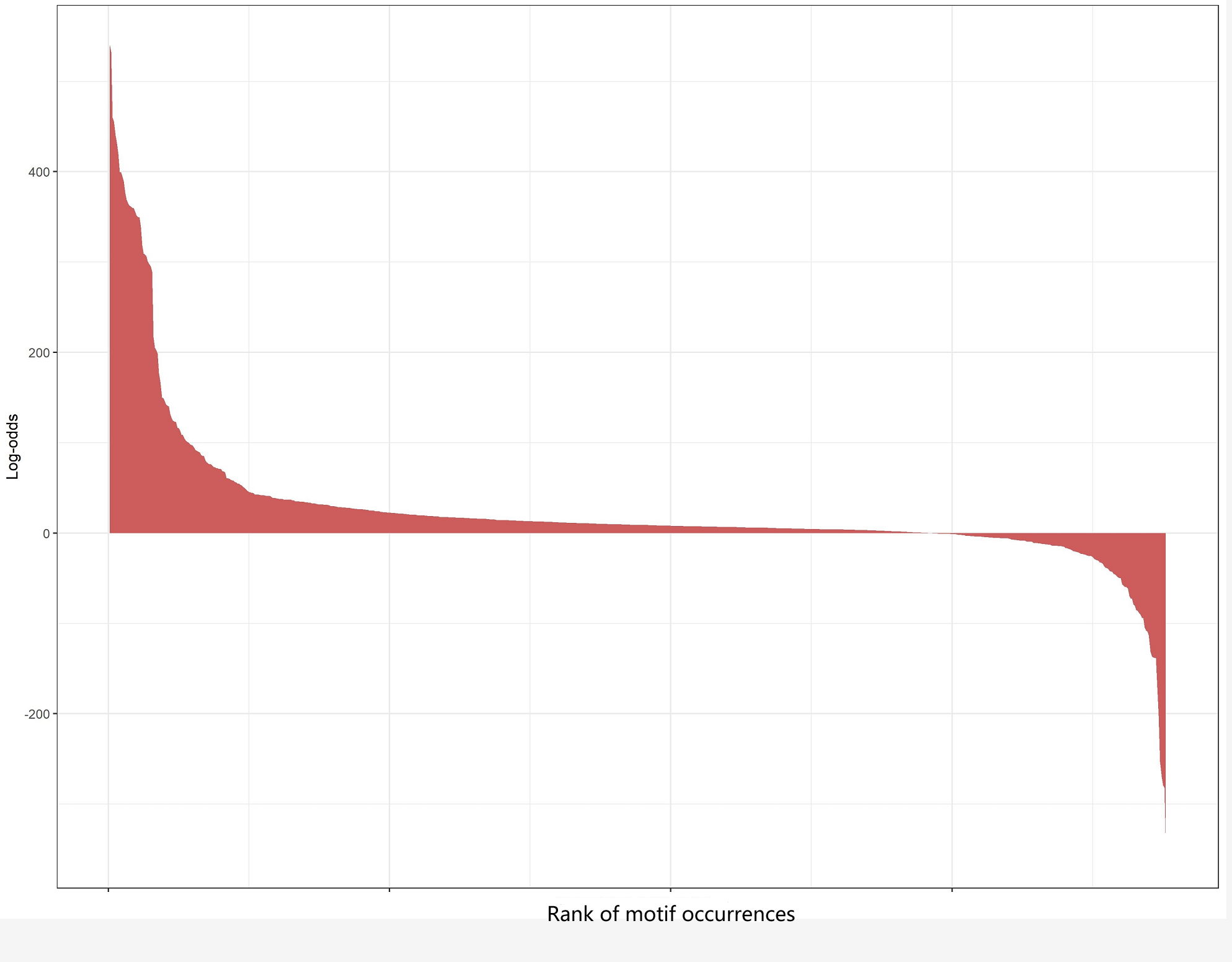
图4:log-odds图。 *注释:由log-odds值对数量化footprinting图中的切割位点
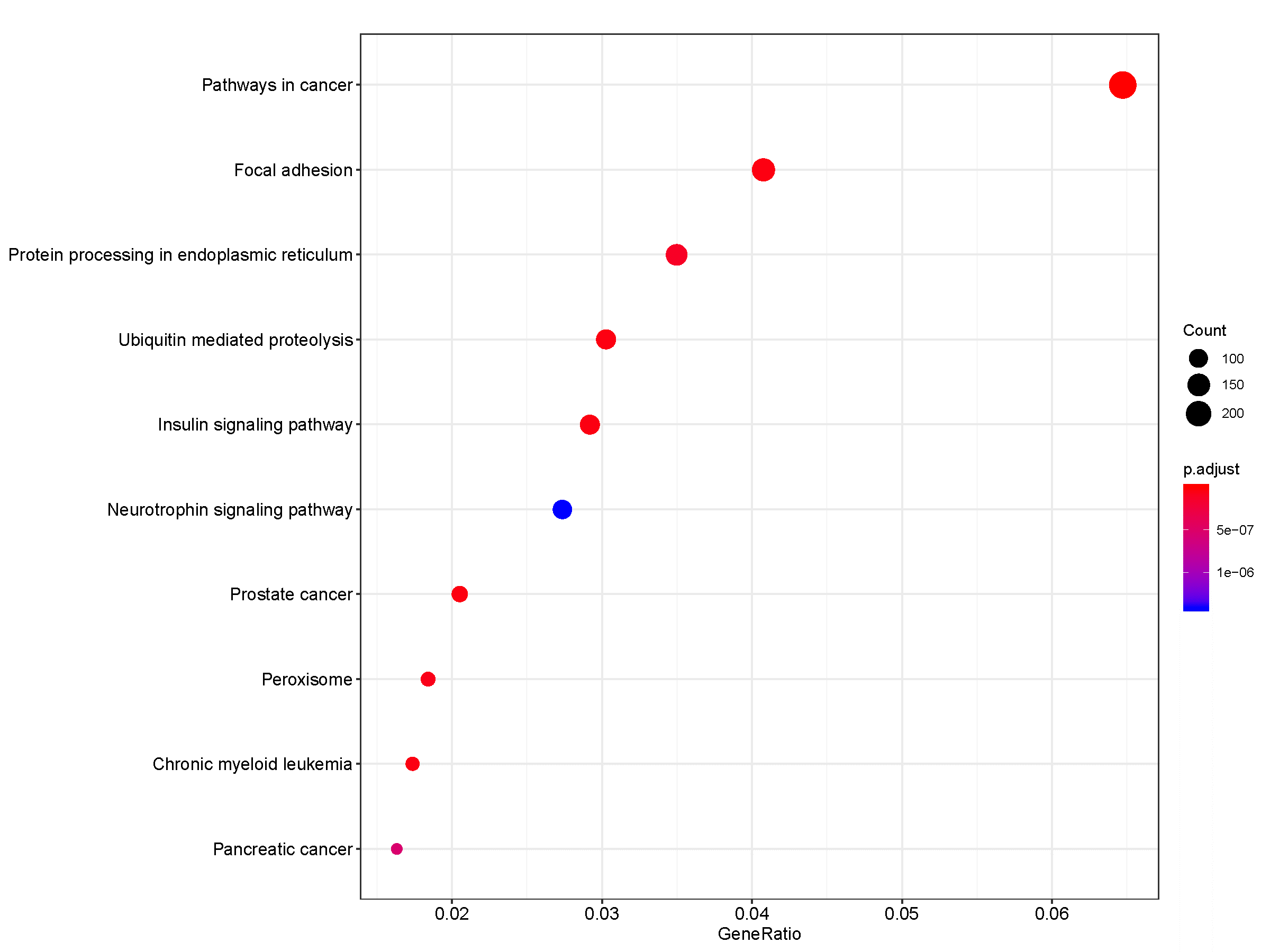
图5:KEGG富集
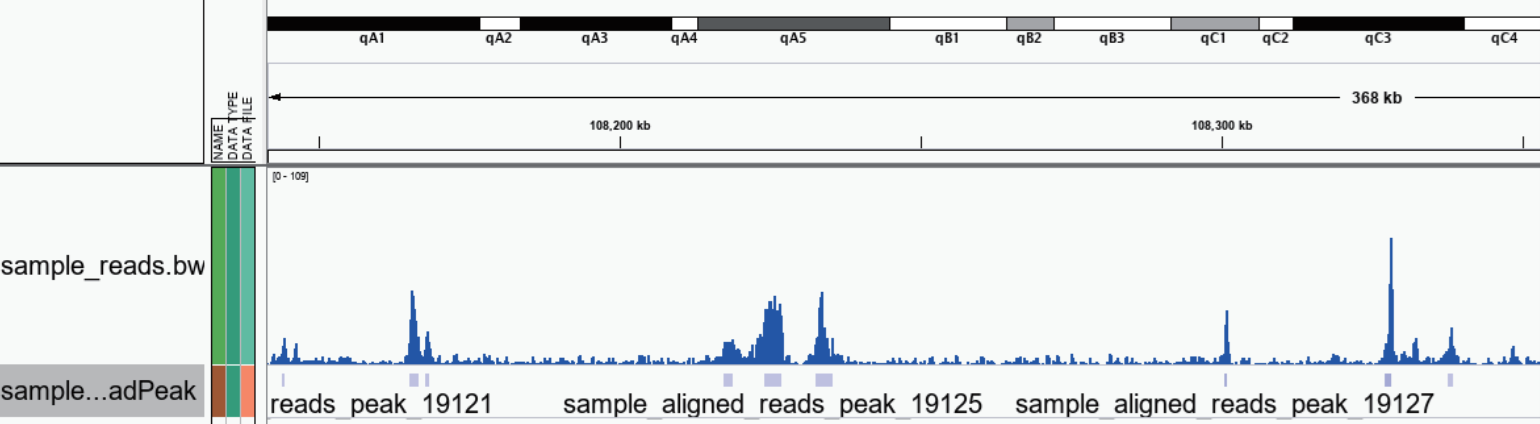
图6:IGV视图

参考案例
CUT & RUN技术的低细胞和高灵敏度的表观基因组学[2]
本研究方法检测多个转录因子(干细胞相关转录因子OCT4、SOX2和NANOG)、组蛋白修饰、鉴定超级增强子、染色质的开放性效果。CUT & RUN技术将蛋白质-DNA相互作用应用于低细胞量研究;甚至在单细胞研究中,仍然有较高的灵敏度。
1. 低细胞用量:
CUT & RUN技术分析CTCF 或H3K4me3测序结果:在这两种蛋白已知结合位点处的read可视化,显示10个细胞起始量的测序结果可以展现出类似50万个细胞量的结合位点富集热图。CTCF的结合区更为狭窄集中,而H3K4me3则占据相对宽的区段。而对照组(无抗体组)的热图则没有这种特征。
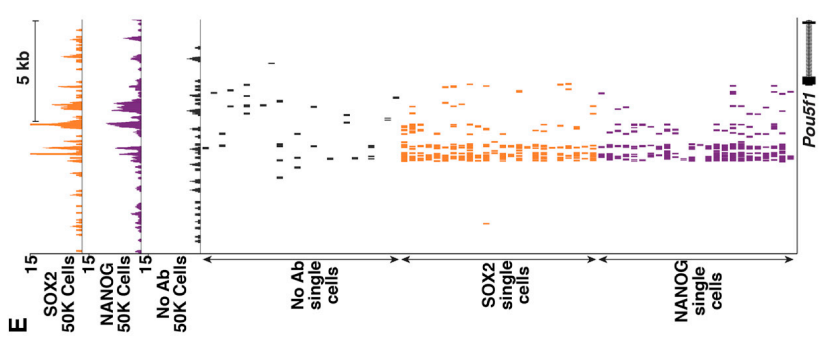
2. 鉴定超级增强子(super-enhancer):
单细胞SOX2和NANOG CUT&RUN数据图中,在它们共同结合的Pou5f1 超级增强子区段内有多处富集。
参考案例
CUT&RUN用于绘制全基因组转录因子结合图谱[3-5]
虽然染色质免疫沉淀(ChIP)技术不断发展,但其DNA富集方法存在缺点却没有很好的改善。CUT & RUN技术作为表观基因组学研究的一种新型技术方法,对传统的方法进行了重大的改进,以消除ChIP固有的缺点。CUT & RUN技术将用于绘制全基因组转录因子结合位点、染色质相关复合物、组蛋白变体和翻译后修饰的图谱;比ChIP更精准高效,其在蛋白质-DNA研究中将会有越来越广泛应用。

CUT & RUN技术检测的信噪比传统ChIP-seq高很多
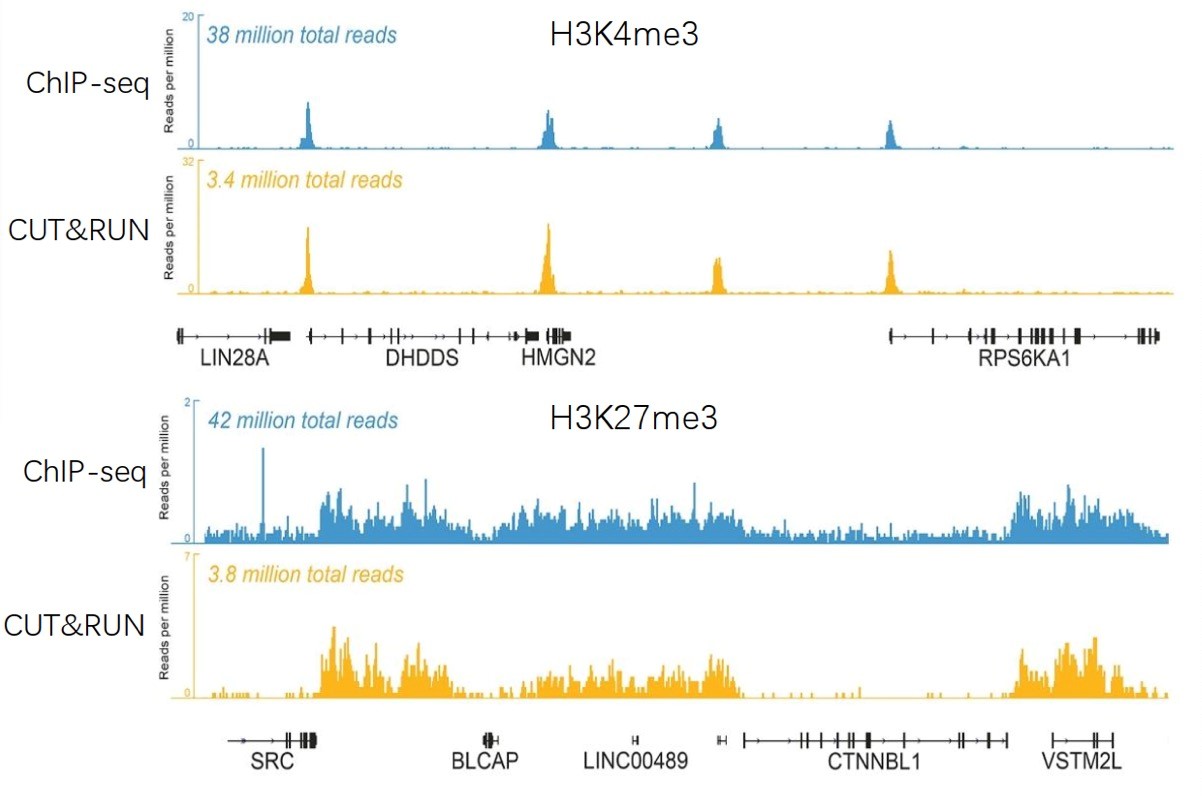
CUT&RUN与ChIP-seq比较
参考文献
[1] Peter J, Steven H. An efficient targeted nuclease strategy for high-resolution mapping of DNA binding sites. Elife, 2017, Jan 16; 6: e21856
[2] Sarah J, Ana B, Kurtis N, et al. Profiling of Pluripotency Factors in Single Cells and Early Embryos. Cell, 2019, May 16; 177: 1-11.
[3] Michael P, Derek H, Steven H. Pioneer factor-nucleosome binding events during differentiation are motif-encoded. Mol Cell, 2019, August 08; 75(3) :562-575.
[4] Skene, Henikoff, Henikoff. Targeted in situ genome-wideprofiling with high efficiency for low cell numbers. Nature protocols, 2018, April 12: 1006-1019.
[5] Meers, Bryson, Henikoff, Henikoff.Improved CUT&RUN chromatin profiling tools. eLife, 2019, Jun 24; 8: e46314.
