CUT&Tag技术服务
项目简介
CUT&Tag技术服务
CUT & Tag(Cleavage Under Targets and Tagmentation),即靶向剪切及转座酶技术,是一种利用酶锚定技术进行高效、高分辨率的DNA测序文库构建方法,适用于蛋白质—DNA互作研究,揭示更多染色质在调控基因表达方面的重要作用,有助于分析细胞量少的样本的特定染色质特征,进一步了解基因调控。Cut & Tag技术革命性地颠覆了以往繁琐复杂的蛋白质—互作研究技术,有助于挖掘以往难以呈现的表观基因组学调控功能,掀起表观遗传学研究领域新的风暴。
技术原理: 将特异的抗体(如H3K27ac),与染色质蛋白原位结合,然后锚定到A-Tn5转座融合蛋白。Tn5转座酶活性被激活,仅将抗体靶蛋白结合的染色质片段化,高效产生高分辨率、低背景的片段文库。
应用与特点
技术应用
1. 挖掘蛋白质—DNA相互作用
2. 检测基因组上的组蛋白修饰,鉴定超级增强子
3. 检测基因组的开放性、转录因子结合图谱,鉴定超级增强子
4. 绘制核小体、RNAPII等蛋白的染色质图谱
技术特点
1. 样本量仅需20万个细胞
2. 操作便捷,一管完成所有步骤
3. 信噪比高(背景低),重复性好
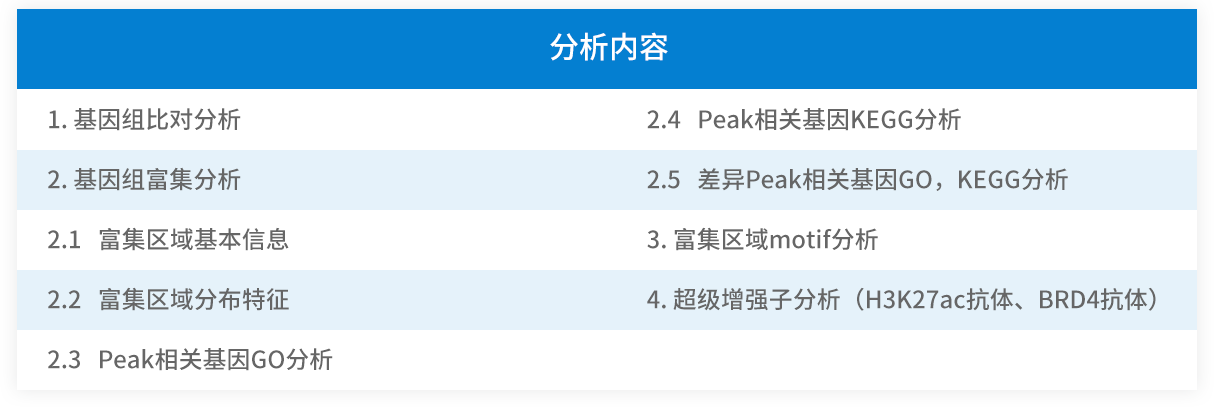
分析内容
送样要求
活细胞
样本类型
2x10^5 ~5 x10^5
细胞数量
人、大小鼠
样本物种
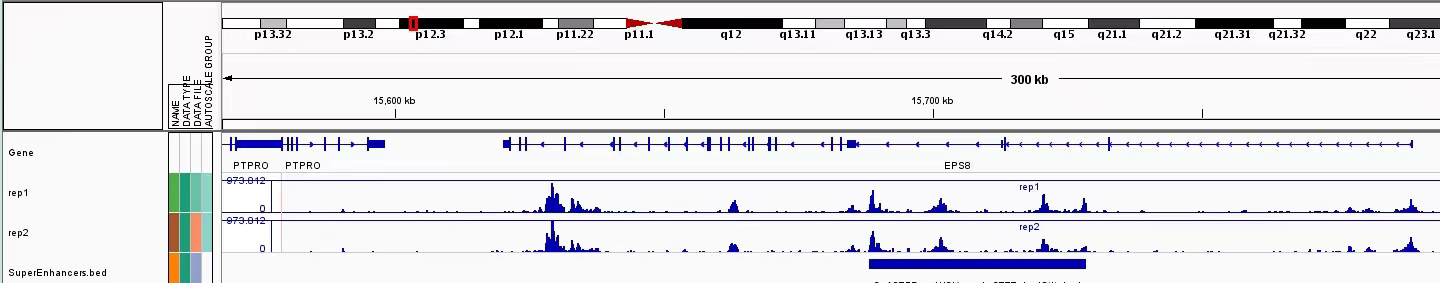
表观生物实测数据 1
表观生物利用CUT&Tag检测组蛋白修饰H3K27me3的读长覆盖图,显示CUT&Tag H3K27me3 peaks(第二行),与文献数据对比(第一行)[1]

表观生物实测数据2
表观生物利用CUT&Tag检测H3K27me3的TSS分布热图(左列),与文献数据对比(右列)[1]
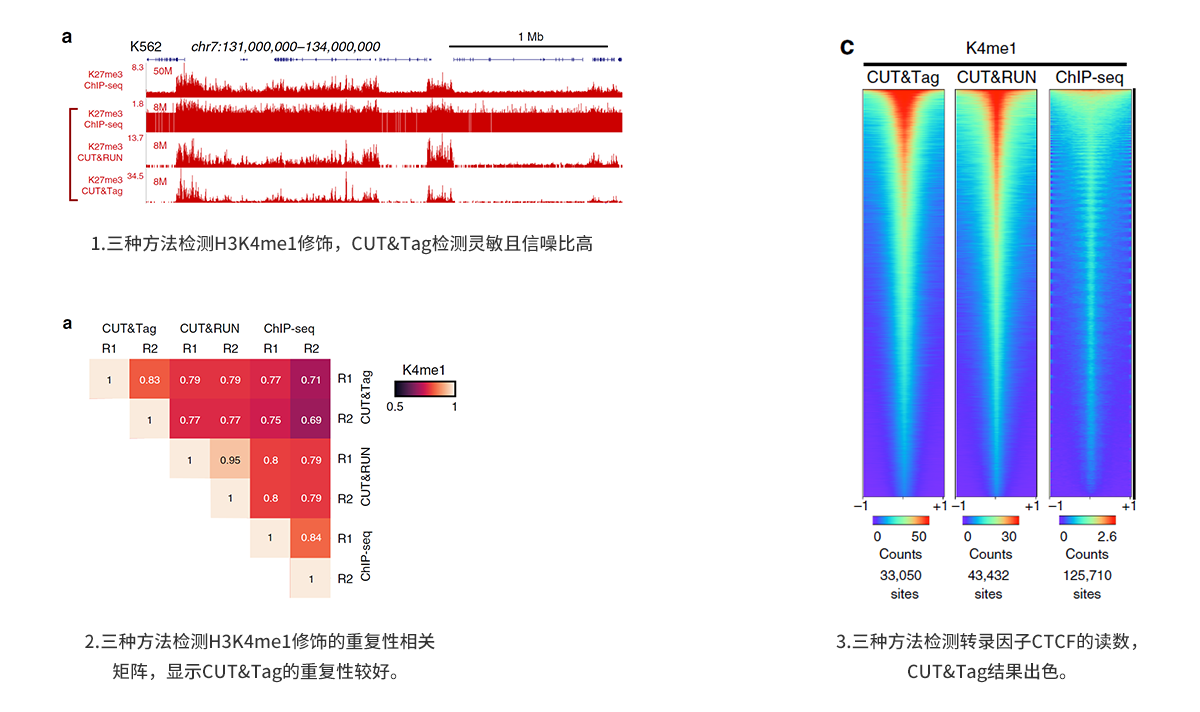
参考案例
Nat Commun:CUT&Tag 高效分析小样本和单细胞的表观基因组学[1]
本研究对比了ChIP-seq、CUT&RUN、CUT&Tag三种方法检测多种组蛋白修饰、RNAPII、转录因子、染色质开放性的效果。通过相同的数据量(8M Reads)进行比较分析发现,三种方法检测结果基本一致,但是ChIP-seq需要更高的测序深度才能达到去背景噪音的效果,而CUT&Tag有最低的背景噪声和最强的信号。
参考文献
[1] Kaya-Okur HS, Wu SJ, Codomo CA, et al. CUT&Tag for efficient epigenomic profiling of small samples and single cells. Nat Commun. 2019 Apr 29;10(1):1930.
